Čekáme na plochou křivku, pojďme si ji zatím nakreslit
Zaslechl jsem zprávu, že cílem karantény a dalších opatření je oploštit křivku. Co tedy dělat, když máme domácí úkoly hotové, látkové roušky ušité a internet pod náporem nadšených homeofficáků kolabuje? Můžeme si kreslit.
Tahle pandemie je skvělá v tom, jak máme takřka real-time data. Nebudu tady hořekovat nad tím, jak jsou nepřesná. Nebo… trochu si tedy zalamentuji:
- každý stát může testovat trochu jinak,
- každý stát zvládá testovat jiné množství lidí,
- některé státy si vyloženě statistiku upravují podle politické situace
- a podobně.
Všude jsou počty nakažených tak trochu podhodnocené, ale v každé zemi v jiné míře. Takže vlastně máme dost přesnou představu pomocí nepřesných čísel. To jen pro začátek, abychom od těchto statistik neměli žádná velká očekávání. Navíc bych chtěl upozornit, že nejsem epidemiolog (což ale lidé v čele této země evidentně také ne).
Nuže k věci. Zahlédl jsem tuhle na Wikipedii krásný obrázek epidemické křivky porovnávající počet nakažených v jednotlivých státech srovnaný ode dne zjištění 100. případu nakažení v dané zemi. Obrázek je bohužel statický a data se každodenně mění, proto by bylo fajn mít něco takového doma a pouštět si to v kormutlivých chvílích těchto dnů.
Získáváme data
Pro tvorbu grafu použijeme R. Aktuální data poskytuje CSSE Johns Hopkins University na GitHubu a jsou dostupná počínaje 22. lednem 2020, kdy už epidemie v Číně běžela naplno. Proto v tomto datasetu Čínu zobrazovat nebudu, v relativním zobrazení by nebyla vypovídající, protože 22. ledna už měla svých 100 případů dávno za sebou (konkrétně 548, jestli se správně dívám). Jsou ovšem i jiné zdroje, jak si ukážeme později.
Začneme tedy načtením balíčků a dat.
library(readr) # read data
library(dplyr) # data manipulation
library(tidyr) # tidy data
library(lubridate) # parse date
library(ggplot2) # charts
data <- read_csv("https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Confirmed.csv")
Upravujeme data
Když se na zdrojová data podíváte, zjistíte, že mají netradiční tvar. Nové dny se nepřipisují dolů (jako položky na účtence), ale doprava. S tím se poněkud hůře pracuje, takže si data převedeme tak, aby byla pod sebou a nikoli vedle sebe. K tomu nám dobře poslouží funkce gather() z balíčku tidyr.
Zbavíme se tedy sloupců Province/State, který obsahuje jen části větších států (Austrálie, USA, Čína a podobně), což my nyní rozlišovat nepotřebujeme, a dále Long a Lat, což jsou souřadnice pro případ, že bychom chtěli dělat mapy. To zatím nechceme. V dalším kroku převedu tabulku tak, aby byla data pod sebou (viz výše), přejmenuji sloupec Country/Region na prosté country a sečtu případy pro dané země (to se hodí právě u těch, které mají statistiky rozdělené podle provincií, zkrátka je sečtu do jedné položky za daný stát).
covid_data <- data %>% select(-"Province/State", -Long, -Lat) %>% gather(date, cases, -"Country/Region") %>% rename(country = "Country/Region") %>% group_by(country, date) %>% summarize(confirmed = sum(cases))
Nyní nám vznikla tabulka se třemi sloupci, tohle jsou první dva řádky:
| country | date | confirmed |
|---|---|---|
| Afghanistan | 1/22/20 | 0 |
| Afghanistan | 1/23/20 | 0 |
Všimněte si hrůzostrašného formátu data. Naštěstí je tady funkce mdy() z balíčku lubridate.
covid_data$date <- covid_data$date %>% mdy()
Teď už tabulka vypadá lépe:
| country | date | confirmed |
|---|---|---|
| Afghanistan | 2020–01–22 | 0 |
| Afghanistan | 2020–01–23 | 0 |
Ještě upravím země, které mě budou zajímat. Data z Taiwanu prošla dramatickým vývojem, přechodnou dobu byla zavzata pod Čínu, proto je u Taiwanu hvězdička. A Jižní Korea je zapsána jako „Korea, South“, což můžeme taky změnit. I některé další země by zasloužily upravit pro potřeby grafu, ale ty teď používat nebudu, takže jen tyto dvě. Použijeme k tomu funkci recode() z balíčku dplyr.
covid_data$country <- covid_data$country %>%
recode(`Taiwan*` = "Taiwan",
`Korea, South` = "South Korea")
A už můžeme vyfiltrovat země, které nás zajímají. Je fajn odstartovat zobrazení na 100. případu, protože úvodní fáze nákazy jsou v zemích různě rozpačité. A vyberu si náhodně země, které mne zajímají.
covid_plot <- covid_data %>%
filter(confirmed > 100 & country %in% c("US", "Taiwan", "Italy", "South Korea", "Czechia", "Singapore", "United Kingdom", "Slovakia", "Spain", "Japan")) %>%
group_by(country) %>%
mutate(days100 = as.numeric(date - min(date)))
Chci na konec každé řady dát popisek dané země (místo klasické legendy). Proto si odfiltruji poslední záznamy pro danou zemi a jen ty pak oštítkuji
covid_last <- covid_plot %>% slice(which.max(date))
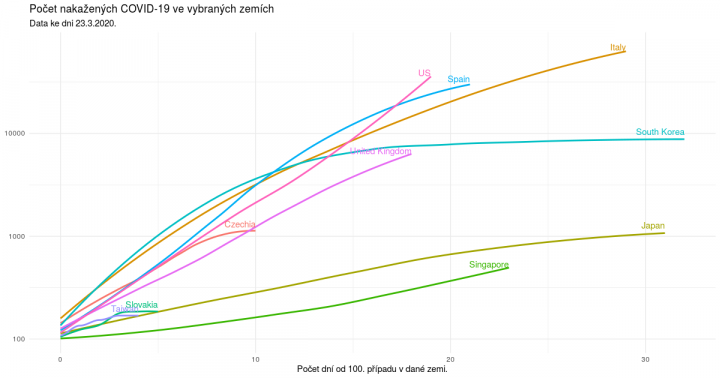
Hurá na graf!
A už můžeme tvořit graf díky balíčku ggplot2. Pro estetický zážitek použiji „vyhlazovací“ funkci geom_smooth(), ale stejně tak by se dala data zobrazit v surovém stavu.
ggplot(covid_plot, aes(days100, confirmed, color = country, label = country)) +
scale_y_log10(breaks = 10^seq(1, 5)) +
geom_smooth(se = FALSE) +
theme_minimal() +
theme(legend.position = "none") +
geom_text(data = covid_last,
hjust = 1, nudge_y = 0.07) +
labs(x = "Počet dní od 100. případu v dané zemi.", y = NULL,
title = "Počet nakažených COVID-19 ve vybraných zemích",
subtitle = paste0("Data ke dni ", format(max(covid_plot$date), "%x"), "."))
Další zdroje dat
Na začátku jsem slíbil, že se podíváme i na jiný zdroj dat. Ten nabízí například balíček nazvaný příznačně nCov19. Je to balíček čínského statistika, takže bohužel tam nenajdete Taiwan. Čísla jsou také lehce odlišná, záleží vždy na zdrojích, odkud autoři čerpají. Balíček v defaultním nastavení stahuje Wuhanský dataset z GitHubu.
Tento kód už nebudu komentovat a vložím jej rovnou s ukázkou grafu:
library(dplyr) # data manipulation
library(ggplot2) # charts
library(nCov2019) # COVID-19 data from https://github.com/canghailan/Wuhan-2019-nCoV
data <- load_nCov2019()
covid_plot <- data["global"] %>%
as_tibble %>%
filter(cum_confirm > 100 & country %in% c("China", "Italy", "South Korea", "Czech Republic", "Singapore", "United Kingdom", "Slovakia", "Spain", "Japan")) %>%
group_by(country) %>%
mutate(days100 = as.numeric(time - min(time)))
covid_last <- covid_plot %>%
slice(which.max(time))
ggplot(covid_plot, aes(days100, cum_confirm, color = country, label = country)) +
scale_y_log10(breaks = 10^seq(1, 4)) +
geom_smooth(se = FALSE) +
theme_minimal() +
theme(legend.position = "none") +
geom_text(data = covid_last,
hjust = 0, nudge_x = 0.2) +
labs(x = "Počet dní od 100. případu v dané zemi.", y = NULL,
title = "Počet nakažených COVID-19 ve vybraných zemích",
subtitle = paste0("Data ke dni ", format(time(data), "%x"), "."))

Všimněte si, že tvar křivek je skutečně trochu jiný. Částečně na tom má podíl „vyhlazování“ křivky, ale data u jednotlivých dnů jsou také trochu odlišná, když oba datasety porovnáme. Celkové vyznění grafu ale zůstává stejné.
A je hotovo
Podobně bychom si mohli sestavit relativní grafy (např. počet nakažených v přepočtu na počet obyvatel), posouvat hranici (např. od 10. nebo od 30. případu nakažení) a podobně. Základ kódu ale zůstane víceméně stejný, pokud jsem tedy někde neudělal chybu, což se může stát. :-)
Takže máme nakresleno, můžeme čekat na oploštění křivky!
Aktualizace
-
Související články na blogu eHealth
-
Máte třetí vlnu? A mohl bych ji vidět? 22. 1. 2021 20:13
-
Vakcína na 95 % 31. 1. 2021 0:54
-
Jak zabalit kolegům data do balíčku v R 27. 1. 2021 15:59
-
Nenechte se zmást odstíny růžové aneb pozor na pandemické grafy 20. 9. 2020 19:25
-
Analýza úniků zdravotnických dat v USA: přibývá vzdálených útoků po síti 2. 10. 2018 23:56
-
Jak je to s tou Mirkou Spáčilovou a jejími 60 % 13. 8. 2018 23:19
-
-
Související články na serveru Root.cz
-
Postřehy z bezpečnosti: chyby v prohlížečích a nová quishingová kampaň 2. 9. 2024 0:00
-
Postřehy z bezpečnosti: Trump's Dumps a další fóra s karetními údaji jsou mimo provoz 21. 2. 2022 0:00
-
Postřehy z bezpečnosti: Apache neví, že není tečka jako tečka 11. 10. 2021 0:00
-
Postřehy z bezpečnosti: kryptoměny táhnou 11. 1. 2021 0:00
-
Co nás čeká v roce 2021: linuxové distribuce, desktopy i hardware 8. 1. 2021 0:00
-
Co se v roce 2020 nepovedlo: šindelové disky, upadající Firefox a ztrácející Intel 29. 12. 2020 0:00
-
-

+1 za "hrůzostrašného formátu data" :)
Ale poprosil bych, nevyhlazovat data. Anebo pokud se data vyhlazují, tak to aspoň vždy uvést do popisku grafu. Vyhlazováním dat ztrácíte informace, a někteří lidé, méně zběhlí v analýze dat a čtení grafů si mohou vytvořit mylný dojem. Samozřejmě někdy je potřeba vybrat jen vhodnou informaci k zobrazení, ale mělo by to být okomentované v grafu.
-

Heh, MJD znam. Pocet dnu od nejakeho data je celkem v pohode. Vlastne na vypocty tahnouci se pres nekolik let to je celkem dobre. Ale poradi mesic-den-rok, z toho mam osypky. A hlavne kdyz to pouzije nekdo jiny nez american. Zrovna dva dny zpatky nejaka prezentace naseho ministerstva zdravotnictvi. Brrrrr.
-

Nejsem tak zběhlý v R, takže úplně nechápu tuhle %>% notaci.
Tedy, tohle je mi jasné:
covid_data$date = covid_data$date %>%
mdy()Odpovídá:
covid_data$date <- mdy(covid_data$date)Ale co si mám představit pod tímhle: ?
covid_data <- data %>%
select(-"Province/State", -Long, -Lat) %>%
gather(date, cases, -"Country/Region") %>%
rename(country = "Country/Region") %>%
group_by(country, date) %>%
summarize(confirmed = sum(cases))Mohl by to někdo prosím přepsat do více srozumitelné formy?
-
Smazaný profil
@PH:
%>%je pipe operator, vizte https://cran.r-project.org/web/packages/magrittr/magrittr.pdf . Vlastně přebírá výsledek předchozího a vkládá jej jako první parametr do další funkce. Něco jako unixová pipeline. -

Hmm tak to tedy asi opravdu funguje,
covid_data <- summarize (group_by(rename(gather(select( data,-"Province/State", -Long, -Lat),date, cases, -"Country/Region"),country = "Country/Region"),country, date),confirmed = sum(cases))
Ale mam z toho %>% zpusobu zapisu takovy ambivalentni pocit, vypada to prehledne, usporne, ale mam obavu, ze v pripade chyby to nedokazu rozklicovat.
Obdobne to mam s temi + na konci radku u ggplot.
Jinak diky za pekny priklad a zdroj dat pro aktualni situaci...
-

Ten operátor není přímo z R. Je z knihovny magrittr a ta je zase součástí Tidyverse, stejně jako ggplot2 nebo knihovny pro manipulaci s daty dplyr a tidyr. Tidyverse představuje poměrně specifický přístup k práci s daty v R a ne každému se líbí.
Mně to smysl dává. Líbí se mi funkcionální nástroje, zpracování dat v několika krocích pomocí pipeline, spojování komponent pro definici grafu. Ale je to další vrstva, se kterou se člověk musí trochu učit.
-

Ahoj!
Hej díky, sám jsem si zpočátku dělal něco podobného v Matlabu, ale data jsem ručně přepisoval (dokud to bylo jen v Číně, a dokud to nezkazili tím, že skokově změnili způsob reportování nakažených.)Co to dát jako projekt na github nebo tak někam, aby se případně mohlo zapojit více lidí a rozšiřovat to?
Rád klidně přispěju (budu se tedy muset naučit s Rkem, což něco zabere), ale docela by mne zajímalo:
- výpočet a zobracení aktuálního R pro jednotlivé země v tabulce
- relativní grafy v přepočtu na počet obyvatel
- analýza dopadu hustoty zalidnění
- zohlednění jednotlivých protiopatření a analýza jejich vlivu (ale získat data by bylo asi dost manuální práce)
- atd...Díky,
Randolf -
Smazaný profil
Ahoj, moc díky za zajímavé tipy. :-) Nechal jsem se tím vyprovokovat a díky tomu jsem dnes poprvé v životě nakoukl do Shiny. Během odpoledne jsem z výše uvedeného skriptu vypotil webovou Shiny aplikaci.
Aplikace běží na webu, umožňuje změnit škálu vertikální osy, startovací počet nakažených i zobrazené země: https://petrkajzar.shinyapps.io/covid19/.
-

Tak tak. Vypada to velmi linearne kdyz se to zohledni. Tyhlety exponencialni grafy spis ukazuji exponencialni narusty testovani tak jak se vladam je dari zvysovat. Zde graf pro CR:
https://twitter.com/lzap/status/1241278052367245313?s=21
Zajimalo by me to jak by to vypadalo pro cely svet.
-

tady mas pocet nakazenych a mrtvych na 1 M obyvatel:
https://www.worldometers.info/coronavirus/z toho je videt, jak jsou na tom nektere staty v porovnani s cinou hodne spatne.
-
Smazaný profil
Dobrý den,
díky za tip! Souhrnná data za EU zní jako dobrý nápad, zapracuji to večer nebo zítra, jakmile se k tomu dostanu.
Co se týče relativních čísel na milion obyvatel, hraji si s tím nějakou dobu a nevychází to dobře. Absolutní graf začíná na n-tém nahlášeném případu v dané zemi. Relativní graf přepočtený na milion obyvatel takto začínat nemůže, protože 100. případ v Číně má jinou relativní váhu než 100. případ v ČR. Zkusil jsem tedy hodnoty zobrazovat např. od dosažení 100. případu na milion obyvatel. Nicméně i tak je graf velmi ovlivněný velikostí země, přístupem k testování a dalšími vlivy. A samozřejmě pak Čína vychází prakticky horizontálně a naopak malé státy jako San Marino či Lucembursko (nebo výletní loď) trčí do nebes. Ani u států se vzájemně podobnou velikostí populace to ale graficky moc nesedí a nepomáhají ani hrátky s vertikální osou. Proto jsem se rozhodl že takto zavádějící graf raději nebudu zveřejňovat, protože nedokážu (neumím) zajistit nějaké srozumitelné grafické vyjádření... Relativní čísla je pak lepší vyjádřit tabulkově, což má například https://www.worldometers.info/coronavirus/#countries.
V každém případě děkuji za tipy a za zájem! :-)
-
Smazaný profil
Hotovo, "European Union" je v seznamu: https://petrkajzar.shinyapps.io/covid19/
Ještě jednou díky za tip.
(autorem editována URL adresa)
-

Zdravim
1)
Tu som si vsimol asi bug, ze ked dam zobrazit daily deaths> USA, tak mierka ukazuje iba 400, pricom denne tam padne vyse 2000 ludi,2)
Apropo neviete ci sa da v tom grafe nastavit ( ci to ten R tool dovoluje )aby aj ukazoval sumu, ked sa nastavim na jednotlie stlpce/bary ?3) neviete ako dam print (vsetko ) po stranach ?, lebo vypise mi iba prvych 10 riadkov
covid_data
# A tibble: 17,205 x 3
# Groups: country [185]
country date confirmed
<chr> <chr> <dbl>
1 Afghanistan 1/22/20 0
2 Afghanistan 1/23/20 0
3 Afghanistan 1/24/20 0
4 Afghanistan 1/25/20 0
5 Afghanistan 1/26/20 0
6 Afghanistan 1/27/20 0
7 Afghanistan 1/28/20 0
8 Afghanistan 1/29/20 0
9 Afghanistan 1/30/20 0
10 Afghanistan 1/31/20 0
# … with 17,195 more rowss tymto mi vypise vsetko ale nie po stranach
> options(dplyr.print_max = 1e9)nieco taketo by sa hodilo `covid_data | more`
A.

Petr Kajzar
Nejčtenější články autora
-
Jak je to s tou Mirkou Spáčilovou a jejími 60 %
Přečteno 57 006×
-
Shodan: Vysokým školám dochází toner!
Přečteno 44 536×
-
Chromebook jako primární (a jediný) laptop?
Přečteno 41 372×
-
Apify: taháme data z webu (aneb jak na kalendář ČLK)
Přečteno 36 422×
-
Nenechte se zmást odstíny růžové aneb pozor na pandemické grafy
Přečteno 33 415×
Poslední názory
-
\
Re: Apify: taháme data z webu (aneb jak na kalendář ČLK)
ke článku Apify: taháme data z webu (aneb jak na kalendář ČLK) -
\
Re: Máte třetí vlnu? A mohl bych ji vidět?
ke článku Máte třetí vlnu? A mohl bych ji vidět? -
\
Re: Máte třetí vlnu? A mohl bych ji vidět?
ke článku Máte třetí vlnu? A mohl bych ji vidět? -
\
Re: Máte třetí vlnu? A mohl bych ji vidět?
ke článku Máte třetí vlnu? A mohl bych ji vidět? -
\
Re: Máte třetí vlnu? A mohl bych ji vidět?
ke článku Máte třetí vlnu? A mohl bych ji vidět?