Posílení klasifikace EKG
Název článku vypadá zvláštně, ale jeho cíl bude po krátkém vysvětlení asi zřejmý.
V několika předchozích dílech povídání jsem se zabýval modely pro klasifikací elektrokardiografu (EKG) s využitím 1D a 2D konvolučních sítí. V tomto volně navazujícím článku si vyberu jeden z těchto modelů a pokusím se jeho výsledky vylepšit pomocí tzv. Ensemble Learning Methods.
Všechny kroky budu provádět na stejné datové sadě, tedy PTB-XL ECG dataset.
Jako základní model, na kterém budu všechny pokusy dělat, jsem vybral kombinovaný model X_Y_model. Jen pro připomenutí, jedná se o plně propojené vrstvy pro metadata a 1D konvoluční vrstvy pro EKG signály.
No a uvidím, kam mne čas zavane …
In [1]:
import sys
import os
import ast
import wfdb
import random
import warnings
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import tensorflow as tf
import tensorflow.keras as keras
import sklearn.metrics
from sklearn.model_selection import train_test_split
from sklearn.model_selection import KFold
sns.set_style('darkgrid')
Příprava dat
Zde budu vycházet z přípravných kroků, které jsem již diskutoval v jednom z předchozích článku. Proto zde uvedu pouze potřebné zdrojové kódy, z nichž bude asi postupu dostatečně zřejmý.
Prvním krokem je načtení metadat datové sady:
In [2]:
PATH_TO_DATA = '/kaggle/input/ptb-xl-dataset/ptb-xl-a-large-publicly-available-electrocardiography-dataset-1.0.1/'
ECG_df = pd.read_csv(os.path.join(PATH_TO_DATA, 'ptbxl_database.csv'), index_col='ecg_id')
ECG_df.scp_codes = ECG_df.scp_codes.apply(lambda x: ast.literal_eval(x))
ECG_df.patient_id = ECG_df.patient_id.astype(int)
ECG_df.nurse = ECG_df.nurse.astype('Int64')
ECG_df.site = ECG_df.site.astype('Int64')
ECG_df.validated_by = ECG_df.validated_by.astype('Int64')
SCP_df = pd.read_csv(os.path.join(PATH_TO_DATA, 'scp_statements.csv'), index_col=0)
SCP_df = SCP_df[SCP_df.diagnostic == 1]
def diagnostic_class(scp):
res = set()
for k in scp.keys():
if k in SCP_df.index:
res.add(SCP_df.loc[k].diagnostic_class)
return list(res)
ECG_df['scp_classes'] = ECG_df.scp_codes.apply(diagnostic_class)
ECG_df.head()
Out[2]:
|
patient_id |
age |
sex |
height |
weight |
nurse |
site |
device |
recording_date |
report |
… |
baseline_drift |
static_noise |
burst_noise |
electrodes_problems |
extra_beats |
pacemaker |
strat_fold |
filename_lr |
filename_hr |
scp_classes |
|
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
ecg_id |
|||||||||||||||||||||
|
1 |
15709 |
56.0 |
1 |
NaN |
63.0 |
2 |
0 |
CS-12 E |
1984–11–09 09:17:34 |
sinusrhythmus periphere niederspannung |
… |
NaN |
, I-V1, |
NaN |
NaN |
NaN |
NaN |
3 |
records100/00000/00001_lr |
records500/00000/00001_hr |
[NORM] |
|
2 |
13243 |
19.0 |
0 |
NaN |
70.0 |
2 |
0 |
CS-12 E |
1984–11–14 12:55:37 |
sinusbradykardie sonst normales ekg |
… |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
2 |
records100/00000/00002_lr |
records500/00000/00002_hr |
[NORM] |
|
3 |
20372 |
37.0 |
1 |
NaN |
69.0 |
2 |
0 |
CS-12 E |
1984–11–15 12:49:10 |
sinusrhythmus normales ekg |
… |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
5 |
records100/00000/00003_lr |
records500/00000/00003_hr |
[NORM] |
|
4 |
17014 |
24.0 |
0 |
NaN |
82.0 |
2 |
0 |
CS-12 E |
1984–11–15 13:44:57 |
sinusrhythmus normales ekg |
… |
, II,III,AVF |
NaN |
NaN |
NaN |
NaN |
NaN |
3 |
records100/00000/00004_lr |
records500/00000/00004_hr |
[NORM] |
|
5 |
17448 |
19.0 |
1 |
NaN |
70.0 |
2 |
0 |
CS-12 E |
1984–11–17 10:43:15 |
sinusrhythmus normales ekg |
… |
, III,AVR,AVF |
NaN |
NaN |
NaN |
NaN |
NaN |
4 |
records100/00000/00005_lr |
records500/00000/00005_hr |
[NORM] |
5 rows × 28 columns
A nyní načtení všech EKG křivek:
In [3]:
def load_raw_data(df, sampling_rate, path):
if sampling_rate == 100:
data = [wfdb.rdsamp(os.path.join(path, f)) for f in df.filename_lr]
else:
data = [wfdb.rdsamp(os.path.join(path, f)) for f in df.filename_hr]
data = np.array([signal for signal, meta in data])
return data
sampling_rate = 100
ECG_data = load_raw_data(ECG_df, sampling_rate, PATH_TO_DATA)
ECG_data.shape
Out[3]:
(21837, 1000, 12)
Následuje příprava datové sady X, Y a Z ze zdrojových dat ECG_df a ECG_data:
In [4]:
X = pd.DataFrame(index=ECG_df.index)
X['age'] = ECG_df.age
X.age.fillna(0, inplace=True)
X['sex'] = ECG_df.sex.astype(float)
X.sex.fillna(0, inplace=True)
X['height'] = ECG_df.height
X.loc[X.height < 50, 'height'] = np.nan
X.height.fillna(0, inplace=True)
X['weight'] = ECG_df.weight
X.weight.fillna(0, inplace=True)
X['infarction_stadium1'] = ECG_df.infarction_stadium1.replace({
'unknown': 0,
'Stadium I': 1,
'Stadium I-II': 2,
'Stadium II': 3,
'Stadium II-III': 4,
'Stadium III': 5
}).fillna(0)
X['infarction_stadium2'] = ECG_df.infarction_stadium2.replace({
'unknown': 0,
'Stadium I': 1,
'Stadium II': 2,
'Stadium III': 3
}).fillna(0)
X['pacemaker'] = (ECG_df.pacemaker == 'ja, pacemaker').astype(float)
X.head()
Out[4]:
|
age |
sex |
height |
weight |
infarction_stadium1 |
infarction_stadium2 |
pacemaker |
|
|---|---|---|---|---|---|---|---|
|
ecg_id |
|||||||
|
1 |
56.0 |
1.0 |
0.0 |
63.0 |
0.0 |
0.0 |
0.0 |
|
2 |
19.0 |
0.0 |
0.0 |
70.0 |
0.0 |
0.0 |
0.0 |
|
3 |
37.0 |
1.0 |
0.0 |
69.0 |
0.0 |
0.0 |
0.0 |
|
4 |
24.0 |
0.0 |
0.0 |
82.0 |
0.0 |
0.0 |
0.0 |
|
5 |
19.0 |
1.0 |
0.0 |
70.0 |
0.0 |
0.0 |
0.0 |
In [5]:
Y = ECG_data Y.shape
Out[5]:
(21837, 1000, 12)
In [6]:
COLUMNS = ['NORM', 'MI', 'STTC', 'CD', 'HYP']
Z = pd.DataFrame(0, index=ECG_df.index, columns=COLUMNS, dtype='int')
for i in Z.index:
for k in ECG_df.loc[i].scp_classes:
Z.loc[i, k] = 1
Z.head()
Out[6]:
|
NORM |
MI |
STTC |
CD |
HYP |
|
|---|---|---|---|---|---|
|
ecg_id |
|||||
|
1 |
1 |
0 |
0 |
0 |
0 |
|
2 |
1 |
0 |
0 |
0 |
0 |
|
3 |
1 |
0 |
0 |
0 |
0 |
|
4 |
1 |
0 |
0 |
0 |
0 |
|
5 |
1 |
0 |
0 |
0 |
0 |
Rozdělení vstupních dat na trénovací, validační a testovací sady:
In [7]:
X_train, Y_train, Z_train = X[ECG_df.strat_fold <= 8], ECG_data[X[ECG_df.strat_fold <= 8].index - 1], Z[ECG_df.strat_fold <= 8]
X_valid, Y_valid, Z_valid = X[ECG_df.strat_fold == 9], ECG_data[X[ECG_df.strat_fold == 9].index - 1], Z[ECG_df.strat_fold == 9]
X_test, Y_test, Z_test = X[ECG_df.strat_fold == 10], ECG_data[X[ECG_df.strat_fold == 10].index - 1], Z[ECG_df.strat_fold == 10]
print(f"Trénovací sady: X_train={X_train.shape} Y_train={Y_train.shape} Z_train={Z_train.shape}")
print(f"Validační sady: X_valid={X_valid.shape} Y_valid={Y_valid.shape} Z_valid={Z_valid.shape}")
print(f"Testovací sady: X_test ={X_test.shape} Y_test={Y_test.shape} Z_test={Z_test.shape}")
Trénovací sady: X_train=(17441, 7) Y_train=(17441, 1000, 12) Z_train=(17441, 5)
Validační sady: X_valid=(2193, 7) Y_valid=(2193, 1000, 12) Z_valid=(2193, 5)
Testovací sady: X_test =(2203, 7) Y_test=(2203, 1000, 12) Z_test=(2203, 5)
Příprava na modelování
Pro vyhodnocení všech modelů použiji jednu společnou funkci, která zobrazí confusion matrix a zároveň vypíše klasifikační metriky Precision, Recall a F1-score. Metriky jsou zároveň také výstupem funkce, takže si je můžu schovat pro závěrečné hodnocení.
In [8]:
def model_quality_reporting(test, pred, *, model_name='model', labels=COLUMNS):
def print_confusion_matrix(confusion_matrix, axes, class_label, class_names, fontsize=14):
df_cm = pd.DataFrame(confusion_matrix, index=class_names, columns=class_names)
try:
heatmap = sns.heatmap(df_cm, annot=True, fmt="d", cbar=False, ax=axes)
except ValueError:
raise ValueError("Confusion matrix values must be integers.")
heatmap.yaxis.set_ticklabels(heatmap.yaxis.get_ticklabels(), rotation=0, ha='right', fontsize=fontsize)
heatmap.xaxis.set_ticklabels(heatmap.xaxis.get_ticklabels(), rotation=45, ha='right', fontsize=fontsize)
axes.set_ylabel('True label')
axes.set_xlabel('Predicted label')
axes.set_title("Class - " + class_label)
fig, ax = plt.subplots(1, 5, figsize=(16, 3))
for axes, cfs_matrix, label in zip(ax.flatten(), sklearn.metrics.multilabel_confusion_matrix(test, pred), labels):
print_confusion_matrix(cfs_matrix, axes, label, ["N", "Y"])
fig.tight_layout()
plt.show()
print(sklearn.metrics.classification_report(test, pred, target_names=labels, zero_division=0))
m = sklearn.metrics.classification_report(test, pred, target_names=labels, output_dict=True, zero_division=False)
m = {(model_name, k): m[k] for k in labels}
return pd.DataFrame.from_dict(m, orient='index')
metrics = pd.DataFrame(columns=("precision", "recall", "f1-score", "support"), index=pd.MultiIndex.from_tuples([], names=('model', 'group')))
Výchozí model
Jak jsem se již zmínil v úvodu, budu vycházet z jednoho základního modelu, a sice kombinovaného modelu pro metadata a EKG křivky. Výstupem modelu je vektor pravděpodobností zařazení vzorku do klasifikačních tříd.
In [9]:
def create_model(X_shape, Y_shape, Z_shape, *, name='Primary Model'):
def create_X_model(X, *, units=32, dropouts=0.3):
X = keras.layers.Normalization(axis=-1, name='X_norm')(X)
X = keras.layers.Dense(units, activation='relu', name='X_dense_1')(X)
X = keras.layers.Dropout(dropouts, name='X_drop_1')(X)
X = keras.layers.Dense(units, activation='relu', name='X_dense_2')(X)
X = keras.layers.Dropout(dropouts, name='X_drop_2')(X)
return X
def create_Y_model(X, *, filters=(32, 64, 128), kernel_size=(5, 3, 3), strides=(1, 1, 1)):
f1, f2, f3 = filters
k1, k2, k3 = kernel_size
s1, s2, s3 = strides
X = keras.layers.Normalization(axis=-1, name='Y_norm')(X)
X = keras.layers.Conv1D(f1, k1, strides=s1, padding='same', name='Y_conv_1')(X)
X = keras.layers.BatchNormalization(name='Y_norm_1')(X)
X = keras.layers.ReLU(name='Y_relu_1')(X)
X = keras.layers.MaxPool1D(2, name='Y_pool_1')(X)
X = keras.layers.Conv1D(f2, k2, strides=s2, padding='same', name='Y_conv_2')(X)
X = keras.layers.BatchNormalization(name='Y_norm_2')(X)
X = keras.layers.ReLU(name='Y_relu_2')(X)
X = keras.layers.MaxPool1D(2, name='Y_pool_2')(X)
X = keras.layers.Conv1D(f3, k3, strides=s3, padding='same', name='Y_conv_3')(X)
X = keras.layers.BatchNormalization(name='Y_norm_3')(X)
X = keras.layers.ReLU(name='Y_relu_3')(X)
X = keras.layers.GlobalAveragePooling1D(name='Y_aver')(X)
X = keras.layers.Dropout(0.5, name='Y_drop')(X)
return X
X_inputs = keras.Input(X_shape[1:], name='X_inputs')
Y_inputs = keras.Input(Y_shape[1:], name='Y_inputs')
X = create_X_model(X_inputs)
Y = create_Y_model(Y_inputs, filters=(64, 128, 256), kernel_size=(7, 3, 3))
X = keras.layers.Concatenate(name='Z_concat')([X, Y])
X = keras.layers.Dense(128, activation='relu', name='Z_dense_1')(X)
X = keras.layers.Dense(128, activation='relu', name='Z_dense_2')(X)
X = keras.layers.Dropout(0.5, name='Z_drop_1')(X)
outputs = keras.layers.Dense(Z_shape[-1], activation='sigmoid', name='Z_outputs')(X)
model = keras.Model(inputs=[X_inputs, Y_inputs], outputs=outputs, name=name)
return model
Můžu si tento základní model přeložit a provést jeho trénování na připravených datových sadách.
In [10]:
model01 = create_model(X_train.shape, Y_train.shape, Z_train.shape, name='PrimaryModel')
model01.compile(optimizer='adam', loss='binary_crossentropy', metrics=['binary_accuracy', 'Precision', 'Recall'])
model01.summary()
Model: "PrimaryModel"
__________________________________________________________________________________________________
Layer (type) Output Shape Param # Connected to
==================================================================================================
Y_inputs (InputLayer) [(None, 1000, 12)] 0 []
Y_norm (Normalization) (None, 1000, 12) 25 ['Y_inputs[0][0]']
Y_conv_1 (Conv1D) (None, 1000, 64) 5440 ['Y_norm[0][0]']
Y_norm_1 (BatchNormalizati (None, 1000, 64) 256 ['Y_conv_1[0][0]']
on)
Y_relu_1 (ReLU) (None, 1000, 64) 0 ['Y_norm_1[0][0]']
Y_pool_1 (MaxPooling1D) (None, 500, 64) 0 ['Y_relu_1[0][0]']
Y_conv_2 (Conv1D) (None, 500, 128) 24704 ['Y_pool_1[0][0]']
Y_norm_2 (BatchNormalizati (None, 500, 128) 512 ['Y_conv_2[0][0]']
on)
Y_relu_2 (ReLU) (None, 500, 128) 0 ['Y_norm_2[0][0]']
X_inputs (InputLayer) [(None, 7)] 0 []
Y_pool_2 (MaxPooling1D) (None, 250, 128) 0 ['Y_relu_2[0][0]']
X_norm (Normalization) (None, 7) 15 ['X_inputs[0][0]']
Y_conv_3 (Conv1D) (None, 250, 256) 98560 ['Y_pool_2[0][0]']
X_dense_1 (Dense) (None, 32) 256 ['X_norm[0][0]']
Y_norm_3 (BatchNormalizati (None, 250, 256) 1024 ['Y_conv_3[0][0]']
on)
X_drop_1 (Dropout) (None, 32) 0 ['X_dense_1[0][0]']
Y_relu_3 (ReLU) (None, 250, 256) 0 ['Y_norm_3[0][0]']
X_dense_2 (Dense) (None, 32) 1056 ['X_drop_1[0][0]']
Y_aver (GlobalAveragePooli (None, 256) 0 ['Y_relu_3[0][0]']
ng1D)
X_drop_2 (Dropout) (None, 32) 0 ['X_dense_2[0][0]']
Y_drop (Dropout) (None, 256) 0 ['Y_aver[0][0]']
Z_concat (Concatenate) (None, 288) 0 ['X_drop_2[0][0]',
'Y_drop[0][0]']
Z_dense_1 (Dense) (None, 128) 36992 ['Z_concat[0][0]']
Z_dense_2 (Dense) (None, 128) 16512 ['Z_dense_1[0][0]']
Z_drop_1 (Dropout) (None, 128) 0 ['Z_dense_2[0][0]']
Z_outputs (Dense) (None, 5) 645 ['Z_drop_1[0][0]']
==================================================================================================
Total params: 185997 (726.56 KB)
Trainable params: 185061 (722.89 KB)
Non-trainable params: 936 (3.66 KB)
__________________________________________________________________________________________________
In [11]:
MODEL_CHECKPOINT = '/kaggle/working/model/model01.ckpt'
callbacks_list = [
keras.callbacks.EarlyStopping(monitor='val_binary_accuracy', patience=10),
keras.callbacks.ModelCheckpoint(filepath=MODEL_CHECKPOINT, monitor='val_binary_accuracy', save_best_only=True)
]
history = model01.fit([X_train, Y_train], Z_train, epochs=100, batch_size=32, callbacks=callbacks_list, validation_data=([X_valid, Y_valid], Z_valid), verbose=False)
model01 = keras.models.load_model(MODEL_CHECKPOINT)
A takto vypadal průběh trénování z pohledu ztrátové funkce a metrik:
In [12]:
sns.relplot(data=pd.DataFrame(history.history), kind='line', height=4, aspect=4) plt.show()
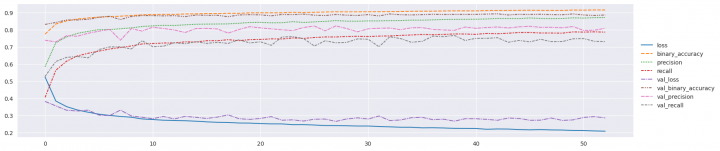
Vyhodnotím si tento základní model. Výsledky mně budou sloužit jako reference pro další pokusy s jeho vylepšením.
In [13]:
Z_pred = np.around(model01.predict([X_test, Y_test], verbose=0)) metrics = pd.concat([metrics.drop(model01.name, level=0, errors='ignore'), model_quality_reporting(Z_test, Z_pred, model_name=model01.name)])
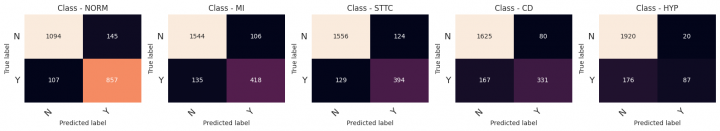
precision recall f1-score support
NORM 0.86 0.89 0.87 964
MI 0.80 0.76 0.78 553
STTC 0.76 0.75 0.76 523
CD 0.81 0.66 0.73 498
HYP 0.81 0.33 0.47 263
micro avg 0.81 0.75 0.78 2801
macro avg 0.81 0.68 0.72 2801
weighted avg 0.81 0.75 0.77 2801
samples avg 0.79 0.76 0.76 2801
První pokus – Model Ensemble
Podstatou vylepšení je vytvoření několika stejných modelů na stejné datové sadě. Predikce ze všech modelů se pak spojí do jedné společné.
Jak by to mělo pomoci?
Všechny váhy modelu jsou v počátečním stavu inicializovány náhodnými hodnotami. Trénováním se pak váhy upravují tak, aby bylo dosaženo co nejmenší odchylky výsledků modelu od očekávaných hodnot pro každý vzorek. Jinak řečeno, každý model bude mít váhy po trénování poněkud rozdílné. Toto by mělo přispět k nižší variabilitě výsledků predikce, možná také ke zlepšení některých metrik.
V tomto pokusu vytvořím 5 modelů, které budu postupně trénovat na stejné datové sadě. Při predikci pak ještě vyzkouším tři varianty, jak spojit výsledky všech modelů do jednoho společného.
In [14]:
ENSEMBLE_SIZE = 5
MODEL_NAME = "ModelEnsemble"
model_ensemble_01 = []
for i in range(ENSEMBLE_SIZE):
print(f"Fitting model instance #{i} ...")
model = create_model(X_train.shape, Y_train.shape, Z_train.shape, name=f'{MODEL_NAME}_{i}')
model.compile(optimizer='adam', loss='binary_crossentropy', metrics=['binary_accuracy', 'Precision', 'Recall'])
MODEL_CHECKPOINT = f'/kaggle/working/model/ensemble_01_{i}.ckpt'
callbacks_list = [
keras.callbacks.EarlyStopping(monitor='val_binary_accuracy', patience=10),
keras.callbacks.ModelCheckpoint(filepath=MODEL_CHECKPOINT, monitor='val_binary_accuracy', save_best_only=True)
]
model.fit([X_train, Y_train], Z_train, epochs=100, batch_size=32, callbacks=callbacks_list, validation_data=([X_valid, Y_valid], Z_valid), verbose=False)
model_ensemble_01.append(keras.models.load_model(MODEL_CHECKPOINT))
Fitting model instance #0 ...
Fitting model instance #1 ...
Fitting model instance #2 ...
Fitting model instance #3 ...
Fitting model instance #4 ...
Nyní mám pět modelů vytrénovaných na stejné datové sadě. Vytvořím si tedy seznam predikcí testovací sady každým modelem zvláště:
In [15]:
predictions = [model.predict([X_test, Y_test], verbose=0) for model in model_ensemble_01]
Jak ale spojit těchto pět predikcí do jedné společné?
Nejdříve zkusím asi ten nejjednodušší postup, a sice průměr pravděpodobností ze všech modelů pro každou klasifikační třídu. Vypíšu si i klasifikační zprávu, abych viděl metriky pro tuto variantu:
In [16]:
pred = np.round(np.average(np.stack(predictions, axis=0), axis=0)).astype(int)
print(sklearn.metrics.classification_report(Z_test, pred, target_names=COLUMNS, zero_division=0))
precision recall f1-score support
NORM 0.84 0.89 0.87 964
MI 0.89 0.69 0.78 553
STTC 0.76 0.78 0.77 523
CD 0.85 0.65 0.74 498
HYP 0.74 0.44 0.56 263
micro avg 0.83 0.74 0.78 2801
macro avg 0.82 0.69 0.74 2801
weighted avg 0.83 0.74 0.78 2801
samples avg 0.79 0.76 0.76 2801
Jako druhou variantu vyzkouším kvorum výsledků modelů. Mám celkem predikce z pěti modelů. Vzorek zařadím do vybrané třídy, pokud jej tak klasifikuje prostá většina ze všech modelů, tedy tři:
In [17]:
pred = (np.sum(np.round(np.stack(predictions, axis=-1)), axis=-1) >= 3.0).astype(int)
print(sklearn.metrics.classification_report(Z_test, pred, target_names=COLUMNS, zero_division=0))
precision recall f1-score support
NORM 0.84 0.89 0.86 964
MI 0.88 0.69 0.77 553
STTC 0.75 0.77 0.76 523
CD 0.86 0.65 0.74 498
HYP 0.73 0.45 0.56 263
micro avg 0.82 0.74 0.78 2801
macro avg 0.81 0.69 0.74 2801
weighted avg 0.82 0.74 0.78 2801
samples avg 0.79 0.76 0.76 2801
V případě klasifikačních úloh ve zdravotnictví bývá při posuzování úspěchu dávána větší váha na výsledky metriky Recall. Je to proto, že je obvykle méně problematické zařazení vzorku tzv. „chybně pozitivní“ než „chybně negativní“. Z tohoto důvodu vyzkouším ještě kvorum s nižším počtem požadovaných pozitivních predikci. Takto to vypadá pro výsledky s hladinou 2:
In [18]:
pred = (np.sum(np.round(np.stack(predictions, axis=-1)), axis=-1) >= 2.0).astype(int)
print(sklearn.metrics.classification_report(Z_test, pred, target_names=COLUMNS, zero_division=0))
precision recall f1-score support
NORM 0.82 0.91 0.86 964
MI 0.86 0.71 0.78 553
STTC 0.73 0.80 0.76 523
CD 0.81 0.69 0.75 498
HYP 0.72 0.51 0.59 263
micro avg 0.80 0.77 0.79 2801
macro avg 0.79 0.72 0.75 2801
weighted avg 0.80 0.77 0.78 2801
samples avg 0.79 0.79 0.78 2801
Pro další posuzování všech pokusů o vylepšení modelů budu dále používat metodu vyhodnocení jako kvorum s hladinou dvě pozitivní predikce.
In [19]:
Z_pred = (np.sum(np.round(np.stack(predictions, axis=-1)), axis=-1) >= 2.0).astype(int) metrics = pd.concat([metrics.drop(MODEL_NAME, level=0, errors='ignore'), model_quality_reporting(Z_test, Z_pred, model_name=MODEL_NAME)])
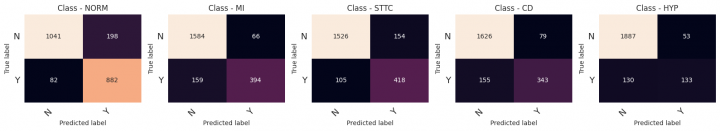
precision recall f1-score support
NORM 0.82 0.91 0.86 964
MI 0.86 0.71 0.78 553
STTC 0.73 0.80 0.76 523
CD 0.81 0.69 0.75 498
HYP 0.72 0.51 0.59 263
micro avg 0.80 0.77 0.79 2801
macro avg 0.79 0.72 0.75 2801
weighted avg 0.80 0.77 0.78 2801
samples avg 0.79 0.79 0.78 2801
Druhý pokus – Random Split Ensemble
Základem pokusu je vytvoření souboru modelů (v mém případě použiji opět 5 modelů). Každý model budu trénovat na náhodně vybrané množině vzorků. Cílem je vytrénovat soubor modelů, které budou na sobě co nejméně závislé. To by mělo přispět ke snížení variability predikce.
Pro pokus budu potřebovat spojit vzorky z množiny X_train a Y_train, se vzorky množiny X_valid a Y_valid. A z takto jedné velké množiny pak náhodně vybírat vzorky pro trénování a validaci.
In [20]:
X_pool = np.vstack([X_train, X_valid])
Y_pool = np.vstack([Y_train, Y_valid])
Z_pool = np.vstack([Z_train, Z_valid])
print(f"{X_pool.shape} {Y_pool.shape} {Z_pool.shape}")
(19634, 7) (19634, 1000, 12) (19634, 5)
Nyní vytvořím postupně pět stejných modelů. Každý pak budu trénovat na náhodně vybraných množinách trénovacích a validačních vzorků z celé sady dat v poměru 4:1.
In [21]:
ENSEMBLE_SIZE = 5
MODEL_NAME = "RandomSplitEnsemble"
model_ensemble_02 = []
for i in range(ENSEMBLE_SIZE):
print(f"Fitting model instance #{i} ...")
model = create_model(X_pool.shape, Y_pool.shape, Z_pool.shape, name=f'{MODEL_NAME}_{i}')
model.compile(optimizer='adam', loss='binary_crossentropy', metrics=['binary_accuracy', 'Precision', 'Recall'])
train_x, valid_x, train_y, valid_y, train_z, valid_z = train_test_split(X_pool, Y_pool, Z_pool, test_size=0.20)
MODEL_CHECKPOINT = f'/kaggle/working/model/ensemble_02_{i}.ckpt'
callbacks_list = [
keras.callbacks.EarlyStopping(monitor='val_binary_accuracy', patience=10),
keras.callbacks.ModelCheckpoint(filepath=MODEL_CHECKPOINT, monitor='val_binary_accuracy', save_best_only=True)
]
model.fit([train_x, train_y], train_z, epochs=100, batch_size=32, callbacks=callbacks_list, validation_data=([valid_x, valid_y], valid_z), verbose=False)
model_ensemble_02.append(keras.models.load_model(MODEL_CHECKPOINT))
Fitting model instance #0 ...
Fitting model instance #1 ...
Fitting model instance #2 ...
Fitting model instance #3 ...
Fitting model instance #4 ...
Takto vypadají predikce všech modelů pro testovací sadu:
In [22]:
predictions = [model.predict([X_test, Y_test], verbose=0) for model in model_ensemble_02]
Do celkového hodnocení zařadím predikce s kvorem 2:
In [23]:
Z_pred = (np.sum(np.round(np.stack(predictions, axis=-1)), axis=-1) >= 2.0).astype(int) metrics = pd.concat([metrics.drop(MODEL_NAME, level=0, errors='ignore'), model_quality_reporting(Z_test, Z_pred, model_name=MODEL_NAME)])
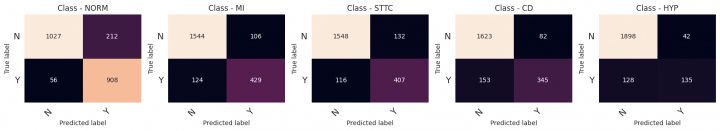
precision recall f1-score support
NORM 0.81 0.94 0.87 964
MI 0.80 0.78 0.79 553
STTC 0.76 0.78 0.77 523
CD 0.81 0.69 0.75 498
HYP 0.76 0.51 0.61 263
micro avg 0.79 0.79 0.79 2801
macro avg 0.79 0.74 0.76 2801
weighted avg 0.79 0.79 0.79 2801
samples avg 0.80 0.80 0.78 2801
Třetí pokus – K-Fold Ensemble
Metoda je variací předchozí Random Split Ensemble. Zde si celou datovou sadu rozdělím na několik stejně velkých bloků. Počet bloků není libovolný, musí odpovídat počtu modelů, které budu chtít vytvořit a trénovat. Pro každý model bude pak jeden z bloků použit jako validační a ostatní jako trénovací, a to tak aby žádný model nepoužíval stejný validační blok.
Mé cíle jsou opět stejné jako v předchozím pokusu, a sice zajistit co největší nezávislost modelů.
In [24]:
ENSEMBLE_SIZE = 5
MODEL_NAME = "KFoldEnsemble"
model_ensemble_03 = []
for i, (train_fold, valid_fold) in enumerate(KFold(ENSEMBLE_SIZE).split(X_pool)):
print(f"Fitting model instance #{i} ...")
model = create_model(X_pool.shape, Y_pool.shape, Z_pool.shape, name=f'{MODEL_NAME}_{i}')
model.compile(optimizer='adam', loss='binary_crossentropy', metrics=['binary_accuracy', 'Precision', 'Recall'])
train_x, valid_x, train_y, valid_y, train_z, valid_z = X_pool[train_fold], X_pool[valid_fold], Y_pool[train_fold], Y_pool[valid_fold], Z_pool[train_fold], Z_pool[valid_fold],
MODEL_CHECKPOINT = f'/kaggle/working/model/ensemble_03_{i}.ckpt'
callbacks_list = [
keras.callbacks.EarlyStopping(monitor='val_binary_accuracy', patience=10),
keras.callbacks.ModelCheckpoint(filepath=MODEL_CHECKPOINT, monitor='val_binary_accuracy', save_best_only=True)
]
model.fit([train_x, train_y], train_z, epochs=100, batch_size=32, callbacks=callbacks_list, validation_data=([valid_x, valid_y], valid_z), verbose=False)
model_ensemble_03.append(keras.models.load_model(MODEL_CHECKPOINT))
Fitting model instance #0 ...
Fitting model instance #1 ...
Fitting model instance #2 ...
Fitting model instance #3 ...
Fitting model instance #4 ...
A takto vypadají predikce modelů:
In [25]:
predictions = [model.predict([X_test, Y_test], verbose=0) for model in model_ensemble_03]
Pokus zakončím zařazením jeho výsledků do celkového přehledu s aplikací kvora 2:
In [26]:
Z_pred = (np.sum(np.round(np.stack(predictions, axis=-1)), axis=-1) >= 2.0).astype(int) metrics = pd.concat([metrics.drop(MODEL_NAME, level=0, errors='ignore'), model_quality_reporting(Z_test, Z_pred, model_name=MODEL_NAME)])
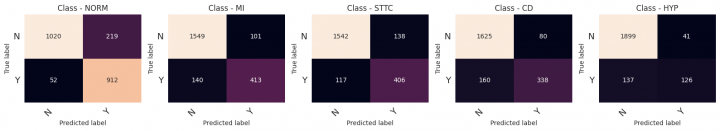
precision recall f1-score support
NORM 0.81 0.95 0.87 964
MI 0.80 0.75 0.77 553
STTC 0.75 0.78 0.76 523
CD 0.81 0.68 0.74 498
HYP 0.75 0.48 0.59 263
micro avg 0.79 0.78 0.79 2801
macro avg 0.78 0.73 0.75 2801
weighted avg 0.79 0.78 0.78 2801
samples avg 0.79 0.80 0.78 2801
Poslední pokus – výběr vzorků s váhou podle zastoupení
Tato varianta je v něčem zvláštní. Pokusím se o vylepšení modelů pro predikci klasifikačních tříd, které jsou v celé datové sadě nejméně zastoupeny. Jistě jste si povšimli, že mám nejhorší výsledky metrik pro klasifikační třídu HYP. Může to pochopitelně být tím, že architektura modelů není schopna tuto třídu rozpoznat. Nabízím ještě jeden možný zdroj problémů s touto třídou:
In [27]:
Z.sum(axis=0)
Out[27]:
NORM 9528 MI 5486 STTC 5250 CD 4907 HYP 2655 dtype: int64
Z výše uvedeného je zřejmé, že v celkové sadě dat mám ty s klasifikací HYP výrazně méně zastoupeny než ty ostatní.
Proto zkusím vytvořit generátor dávek pro trénovaní a validaci, který bude z celkové sady náhodně vybírat vzorky tak, aby byly všechny třídy rovnoměrně zastoupeny. Jinak řečeno, bude výrazně pravděpodobnější zařazení vzorku s klasifikací HYP než například vzorku s klasifikací NORM.
Je zde ještě jeden zádrhel, a sice ten, že jeden vzorek může mít přiřazeno více klasifikačních tříd. V tomto případě jsem se rozhodl dát vzorku pravděpodobnost výběru odpovídající pravděpodobnosti nejméně zastoupené třídy (maximum vah odpovídajícím třídám zastoupeným ve vzorku).
In [28]:
class DataGenerator(keras.utils.Sequence):
def __init__(self, x, y, z, *, batch_size=32, batch_number=None):
self.x = x
self.y = y
self.z = z
self.sample_number = min([self.x.shape[0], self.y.shape[0], self.z.shape[0]])
self.batch_size = batch_size
self.batch_number = batch_number or self.sample_number // self.batch_size
self.class_probabilities = 1 / (z.sum(axis=0) / z.sum())
self.class_probabilities = self.class_probabilities / self.class_probabilities.sum()
@property
def x_shape(self):
return (self.batch_size, ) + self.x.shape[1:]
@property
def y_shape(self):
return (self.batch_size, ) + self.y.shape[1:]
@property
def z_shape(self):
return (self.batch_size, ) + self.z.shape[1:]
def __len__(self):
return self.batch_number
def __getitem__(self, idx):
samples = []
while len(samples) < self.batch_size:
i = random.randrange(self.sample_number)
if (self.class_probabilities * self.z[i]).max() >= random.random():
samples.append(i)
return (self.x[samples], self.y[samples]), self.z[samples]
Jen abyste měli představu, jak ten generátor funguje, malá ukázka vygenerování 10-ti batch a zastoupení klasifikačních tříd v nich:
In [29]:
for (x, y), z in DataGenerator(X_train.to_numpy(), Y_train, Z_train.to_numpy(), batch_number=10):
print(z.sum(axis=0))
[13 10 5 11 5]
[ 9 7 8 10 11]
[11 7 8 12 7]
[14 9 9 7 8]
[ 6 11 9 11 9]
[12 8 7 9 7]
[10 9 11 8 5]
[ 8 9 11 10 7]
[ 7 10 15 6 13]
[10 8 8 14 9]
Vytvořím dva samostatné generátory pro trénování a validaci na oddělených sadách dat. Výsledek bude opět soubor pěti modelů, predikace pak spojena kvorem:
In [30]:
ENSEMBLE_SIZE = 5
MODEL_NAME = "ResamplingEnsemble"
model_ensemble_04 = []
train_gen = DataGenerator(X_train.to_numpy(), Y_train, Z_train.to_numpy())
valid_gen = DataGenerator(X_valid.to_numpy(), Y_valid, Z_valid.to_numpy())
for i in range(ENSEMBLE_SIZE):
print(f"Fitting model instance #{i} ...")
model = create_model(train_gen.x_shape, train_gen.y_shape, train_gen.z_shape, name=f'{MODEL_NAME}_{i}')
model.compile(optimizer='adam', loss='binary_crossentropy', metrics=['binary_accuracy', 'Precision', 'Recall'])
MODEL_CHECKPOINT = f'/kaggle/working/model/ensemble_04_{i}.ckpt'
callbacks_list = [
keras.callbacks.EarlyStopping(monitor='val_binary_accuracy', patience=10),
keras.callbacks.ModelCheckpoint(filepath=MODEL_CHECKPOINT, monitor='val_binary_accuracy', save_best_only=True)
]
model.fit(train_gen, epochs=100, batch_size=32, callbacks=callbacks_list, validation_data=valid_gen, verbose=False)
model_ensemble_04.append(keras.models.load_model(MODEL_CHECKPOINT))
Fitting model instance #0 ...
Fitting model instance #1 ...
Fitting model instance #2 ...
Fitting model instance #3 ...
Fitting model instance #4 ...
Predikace na testovací sadě:
In [31]:
predictions = [model.predict([X_test, Y_test], verbose=0) for model in model_ensemble_04]
A nyní ještě závěrečné vyhodnocení varianty a zařazení výsledků do společného přehledu:
In [32]:
Z_pred = (np.sum(np.round(np.stack(predictions, axis=-1)), axis=-1) >= 2.0).astype(int) metrics = pd.concat([metrics.drop(MODEL_NAME, level=0, errors='ignore'), model_quality_reporting(Z_test, Z_pred, model_name=MODEL_NAME)])
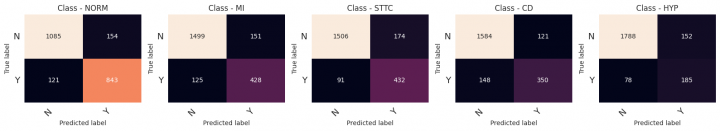
precision recall f1-score support
NORM 0.85 0.87 0.86 964
MI 0.74 0.77 0.76 553
STTC 0.71 0.83 0.77 523
CD 0.74 0.70 0.72 498
HYP 0.55 0.70 0.62 263
micro avg 0.75 0.80 0.77 2801
macro avg 0.72 0.78 0.74 2801
weighted avg 0.75 0.80 0.77 2801
samples avg 0.77 0.80 0.77 2801
Vyhodnocení všech modelů
Pro závěrečné zhodnocení uvedu nejdříve grafy pro metriky Precision, Recall a F1-score.
In [33]:
metrics.index.set_names(['model', 'group'], inplace=True) metrics.reset_index(inplace=True)
Metrika Precision
In [34]:
fig, ax = plt.subplots(figsize=(8, 3)) ax = sns.barplot(metrics, x='group', y='precision', hue='model', ax=ax) ax.set(ylim=(0.2, 1.0)) sns.move_legend(ax, "upper left", bbox_to_anchor=(1, 1)) plt.show()
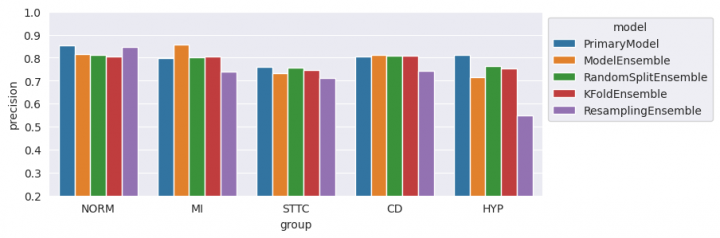
Metrika Recall
In [35]:
fig, ax = plt.subplots(figsize=(8, 3)) ax = sns.barplot(metrics, x='group', y='recall', hue='model', ax=ax) ax.set(ylim=(0.2, 1.0)) sns.move_legend(ax, "upper left", bbox_to_anchor=(1, 1)) plt.show()
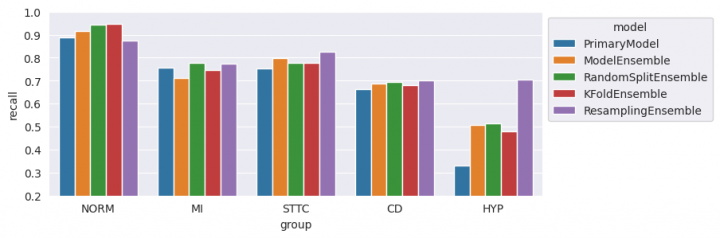
Metrika F1-score
In [36]:
fig, ax = plt.subplots(figsize=(8, 3)) ax = sns.barplot(metrics, x='group', y='f1-score', hue='model', ax=ax) ax.set(ylim=(0.2, 1.0)) sns.move_legend(ax, "upper left", bbox_to_anchor=(1, 1)) plt.show()
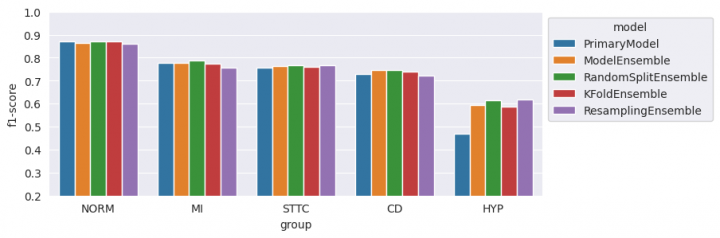
Komentář k výsledkům
Z mého pohledu je podstatný výsledek metriky F1-score. Podle tohoto ukazatele vyšly všechny „Ensemble“ modely o něco málo lépe než primární model s jednou výjimkou, a to je třída HYP. Ta je ve výsledcích výrazně lepší.
Metrika Recall vychází výrazně lépe u třídy HYP, což byl také můj cíl. Za pozornost stojí také horší výsledek u nejčastěji zastoupené třídy NORM.
A poslední zmínka k metrice Precision. Zde vychází všechny „Ensemble“ modely hůře než primární model. To by mohlo být na první pohled zklamání. Vysvětlením je spojením výsledků modelů do jedné predikce. Záměrně jsem zvolil kvorum s hranicí pozitivních klasifikací u nejméně dvou modelů. Cílem tohoto kvóra bylo právě posílení metriky Recall, takže muselo utrpět Precision.
Žádné názory

Jiří Raška
pracuje na pozici IT architekta. Poslední roky se zaměřuje na integrační a komunikační projekty ve zdravotnictví. Mezi jeho koníčky patří také paragliding a jízda na horském kole.
Nejčtenější články autora
-
Mravenčí kolonií na Nurikabe
Přečteno 33 904×
-
AutoEncoder na ořechy
Přečteno 30 321×
-
Klasifikace EKG křivek - výlet do světa neuronových sítí
Přečteno 27 609×
-
Detekce anomálií v auditních záznamech - časové řady
Přečteno 25 519×
-
Detekce anomálií v auditních záznamech
Přečteno 21 002×
Poslední názory
-
\
Re: Detectron2 – hra na špióny
ke článku Detectron2 – hra na špióny -
\
Re: Detectron2 – najdi medvěda
ke článku Detectron2 – najdi medvěda -
\
Re: Mravenčí kolonií na Nurikabe
ke článku Mravenčí kolonií na Nurikabe -
\
Re: AutoEncoder na ořechy
ke článku AutoEncoder na ořechy -
\
Re: Rozpoznání zápalu plic z RTG snímků - ViT model
ke článku Rozpoznání zápalu plic z RTG snímků - ViT model