Lokalizace infekce malárie - klasifikační model a heatmap
Tímto příspěvkem bych chtěl volně navázat na předchozí články zabývající se klasifikací obrázků do několika tříd. V těch dřívějších článcích se jednalo o rozpoznání zápalu plic na základě RTG snímků.
Pokud bych měl rychle shrnout jejich obsah, pak se jednalo o to, že jsem měl k dispozici snímky zdravých plic a snímky se zápalem. Úkolem hledaných modelů bylo najím způsob, jak rozlišit jedny od druhých. Jedná se tedy o běžně studovanou metodu klasifikace snímků na základě dříve připravených vzorků dat (tzv. supervised machine learning).
Otázka je, zda by se z této metody nedalo vytěžit více. Co kdyby mne zajímalo nejen to, že na vybraném snímku je nějaký problém, ale také místo snímku, kde ten problém je. Dalo by se inkriminované místo na snímku najít, aniž bych jej dříve měl označené na trénovacích datech?
Na tyto otázky se pokusím odpovědět v následujících odstavcích článku.
Nejdříve pouze základní příprava prostředí:
import sys import os import shutil import warnings import glob os.environ['TF_CPP_MIN_LOG_LEVEL'] = '3' import numpy as np import pandas as pd import matplotlib.pyplot as plt import seaborn as sns from tqdm import tqdm import tensorflow as tf import tensorflow.keras as keras from keras import Input, Model from keras import layers from sklearn.model_selection import train_test_split from sklearn.metrics import classification_report,confusion_matrix from skimage.feature import peak_local_max from matplotlib.patches import Rectangle import cv2 sns.set_style('darkgrid') warnings.simplefilter(action='ignore', category=FutureWarning)
import random def seed_all(value=42): random.seed(value) np.random.seed(value) tf.random.set_seed(value) os.environ['PYTHONHASHSEED'] = str(value) seed_all()
Datová sada
Pro své pokusy jsem si vybral datovou sadu Malaria Cell Images Dataset. V této datové sadě se dají najít dvě skupiny snímků buněk. První skupina snímků jsou ty, které jsou infikovány malárií. Druhá skupina jsou pak snímky buněk zdravých. Mám tedy k dispozici dvě třídy pro klasifikaci.
Vzhledem k počtu snímků budu všechny načítat jako RGB v menším rozlišení 64×64 bodů.
IMAGE_ROOT = "/kaggle/input/cell-images-for-detecting-malaria/cell_images" LABELS = ['Uninfected', 'Parasitized'] IMAGE_SIZE = (64, 64)
Pro načtení dat do paměti si připravím jednoduchou proceduru. Pro obě sady snímků udělám jejich konverzi do RGB a na požadované rozlišení. Navíc udělám normalizaci hodnot do rozmezí 0 až 1.
Výsledkem procedury jsou dvě pole. První je pole se snímky, druhé pak pole se zařazení snímku do třídu „Uninfected“ nebo „Parasitized“.
Vzhledem k tomu, že vzorky v jednotlivých třídách načítám postupně, zařadil jsem na konec jejich permutaci. To je důležité pro průběh učení, aby byly vzorky ze všech tříd rovnoměrně zastoupeny ve všech dávkách (batch).
def get_data():
x, y = list(), list()
for i, label in enumerate(LABELS):
for fn in tqdm(glob.glob(os.path.join(IMAGE_ROOT, label, '*.png')), desc=label):
image = cv2.imread(fn, flags=cv2.IMREAD_COLOR)
image = cv2.cvtColor(image, cv2.COLOR_BGR2RGB)
image = cv2.resize(image, IMAGE_SIZE).astype(np.float32) / 255.0
x.append(image)
y.append(i)
perm = np.random.permutation(len(x))
return np.array(x)[perm], np.eye(2)[y].astype(np.float32)[perm]
x, y = get_data()
Uninfected: 100%|██████████| 13779/13779 [01:24<00:00, 162.23it/s]
Parasitized: 100%|██████████| 13779/13779 [01:28<00:00, 155.96it/s]
Pro lepší představu o datech je vždy příjemné si zobrazit nějaké vzorky.
Takto vypadají neinfikované vzorky:
samples = x[y[:, 0].astype(np.bool_)] rows, cols = 2, 5 fig=plt.figure(figsize=(14, 6)) for i in range(1, rows*cols+1): fig.add_subplot(rows, cols, i) image = samples[random.randrange(samples.shape[0])] plt.imshow(image) plt.show()
A takto vypadají vzorky infikované malárií:
samples = x[y[:, 1].astype(np.bool_)] rows, cols = 2, 5 fig=plt.figure(figsize=(14, 6)) for i in range(1, rows*cols+1): fig.add_subplot(rows, cols, i) image = samples[random.randrange(samples.shape[0])] plt.imshow(image) plt.show()
Jako obvykle si ještě všechna data rozdělím na dvě skupiny, první pro trénování a druhá pro ověření výsledků modelu:
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2) print(x_train.shape, y_train.shape) print(x_test.shape, y_test.shape) (22046, 64, 64, 3) (22046, 2) (5512, 64, 64, 3) (5512, 2)
Pro trénování modelu mám tedy k dispozici 22046 vzorků v rozlišení 64×64 bodů, se třemi kanály pro kódování barvy. Vzorků pro testování mám k dispozici 5512.
Klasifikační model
Klasifikační model bude víceméně klasický, postavený na konvolučních vrstvách tak, jak je obvyklé ve VGG16 modelech.
Pro zlepšení výsledků učení jsem do modelu doplnil také vrstvy pro úpravu obrázků:
data_augmentation = keras.Sequential([
layers.Normalization(),
layers.RandomFlip("horizontal"),
layers.RandomRotation(factor=0.2),
layers.RandomZoom(height_factor=0.2, width_factor=0.2),
], name="data_augmentation")
data_augmentation.layers[0].adapt(x_train)
Takto vypadá funkce, která mi vytvoří model v základní podobě:
def create_model(X_shape, classes=2, name="Model"):
def conv_block(x, filters, *, kernels=None, steps=None, pooling=True, name=""):
for i in range(len(filters)):
x = layers.Conv2D(filters[i], kernels[i] if kernels else (3, 3), strides=steps[i] if steps else (1, 1), padding='same', name=f'{name}_conv_{i}')(x)
x = layers.BatchNormalization(name=f'{name}_norm_{i}')(x)
x = layers.Activation('relu', name=f'{name}_relu_{i}')(x)
if pooling:
x = layers.MaxPooling2D((2, 2), name=f'{name}_maxpool')(x)
return x
inputs = x = Input(X_shape[-3:], name='inputs')
x = data_augmentation(inputs)
x = conv_block(x, (64, 64), name="block_1")
x = conv_block(x, (128, 128), name="block_2")
x = conv_block(x, (256, 256), pooling=False, name="block_3")
x = keras.layers.GlobalAveragePooling2D(name="global_average_pooling")(x)
outputs = layers.Dense(classes, activation='softmax', name='outputs')(x)
return Model(inputs=inputs, outputs=outputs, name=name)
Model je složen ze tří konvolučních bloků, které obsahují dvě konvoluční vrstvy následované vrstvou MaxPooling2D pro redukci prostorových dimenzí. Doposud tedy nic nového.
Ve VGG16 modelu by po konvolučních blocích obvykle následovala vrstva Flatten. V mém případě je ale nahrazena vrstvou GlobalAveragePoolong2D. Výsledkem této vrstvy je úplné potlačení prostorových dimenzí, zůstanou pouze průměrné hodnoty vlastností.
Celý model je zakončen jednou Dense vrstvou, která dělá rozlišení do jednotlivých tříd.
Pokud se podíváte do výpisu přeloženého modelu uvidíte, že jsem se postupně ze vzorků 64×64×3 konvolučními bloky dostal do vzorku 16×16×256. Odtud pak GlobalAveragePooling2D vrstva vybere průměry vlastností, tedy 256. Ty jsou pak vstupem do Dense vrstvy.
EPOCHS = 20 BATCH_SIZE = 64 model = create_model(x.shape, 2) model.compile(optimizer="adam", loss='categorical_crossentropy', metrics=['accuracy']) model.summary(expand_nested=True) MODEL_CHECKPOINT = f"/kaggle/working/model/{model.name}.keras" callbacks_list = [ keras.callbacks.EarlyStopping(monitor='val_accuracy', patience=10), keras.callbacks.ModelCheckpoint(filepath=MODEL_CHECKPOINT, monitor='val_accuracy', save_best_only=True, mode='max', verbose=1) ] history = model.fit(x=x_train, y=y_train, epochs=EPOCHS, batch_size=BATCH_SIZE, callbacks=callbacks_list, validation_split=0.2, verbose=1) model.load_weights(MODEL_CHECKPOINT) Model: "Model" ┏━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━┳━━━━━━━━━━━━━━━━━━━━━━━━┳━━━━━━━━━━━━━━━┓ ┃ Layer (type) ┃ Output Shape ┃ Param # ┃ ┡━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╇━━━━━━━━━━━━━━━━━━━━━━━━╇━━━━━━━━━━━━━━━┩ │ inputs (InputLayer) │ (None, 64, 64, 3) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ data_augmentation (Sequential) │ (None, 64, 64, 3) │ 7 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ └ normalization │ (None, 64, 64, 3) │ 7 │ │ (Normalization) │ │ │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ └ random_flip (RandomFlip) │ (None, 64, 64, 3) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ └ random_rotation │ (None, 64, 64, 3) │ 0 │ │ (RandomRotation) │ │ │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ └ random_zoom (RandomZoom) │ (None, 64, 64, 3) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_1_conv_0 (Conv2D) │ (None, 64, 64, 64) │ 1,792 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_1_norm_0 │ (None, 64, 64, 64) │ 256 │ │ (BatchNormalization) │ │ │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_1_relu_0 (Activation) │ (None, 64, 64, 64) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_1_conv_1 (Conv2D) │ (None, 64, 64, 64) │ 36,928 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_1_norm_1 │ (None, 64, 64, 64) │ 256 │ │ (BatchNormalization) │ │ │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_1_relu_1 (Activation) │ (None, 64, 64, 64) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_1_maxpool (MaxPooling2D) │ (None, 32, 32, 64) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_2_conv_0 (Conv2D) │ (None, 32, 32, 128) │ 73,856 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_2_norm_0 │ (None, 32, 32, 128) │ 512 │ │ (BatchNormalization) │ │ │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_2_relu_0 (Activation) │ (None, 32, 32, 128) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_2_conv_1 (Conv2D) │ (None, 32, 32, 128) │ 147,584 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_2_norm_1 │ (None, 32, 32, 128) │ 512 │ │ (BatchNormalization) │ │ │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_2_relu_1 (Activation) │ (None, 32, 32, 128) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_2_maxpool (MaxPooling2D) │ (None, 16, 16, 128) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_3_conv_0 (Conv2D) │ (None, 16, 16, 256) │ 295,168 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_3_norm_0 │ (None, 16, 16, 256) │ 1,024 │ │ (BatchNormalization) │ │ │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_3_relu_0 (Activation) │ (None, 16, 16, 256) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_3_conv_1 (Conv2D) │ (None, 16, 16, 256) │ 590,080 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_3_norm_1 │ (None, 16, 16, 256) │ 1,024 │ │ (BatchNormalization) │ │ │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ block_3_relu_1 (Activation) │ (None, 16, 16, 256) │ 0 │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ global_average_pooling │ (None, 256) │ 0 │ │ (GlobalAveragePooling2D) │ │ │ ├─────────────────────────────────┼────────────────────────┼───────────────┤ │ outputs (Dense) │ (None, 2) │ 514 │ └─────────────────────────────────┴────────────────────────┴───────────────┘ Total params: 1,149,513 (4.39 MB) Trainable params: 1,147,714 (4.38 MB) Non-trainable params: 1,799 (7.03 KB) Epoch 1/20 276/276 ━━━━━━━━━━━━━━━━━━━━ 0s 61ms/step - accuracy: 0.8318 - loss: 0.3889 Epoch 1: val_accuracy improved from -inf to 0.76825, saving model to /kaggle/working/model/Model.keras 276/276 ━━━━━━━━━━━━━━━━━━━━ 30s 70ms/step - accuracy: 0.8321 - loss: 0.3884 - val_accuracy: 0.7683 - val_loss: 0.4583 Epoch 2/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 57ms/step - accuracy: 0.9440 - loss: 0.1675 Epoch 2: val_accuracy improved from 0.76825 to 0.93515, saving model to /kaggle/working/model/Model.keras 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 61ms/step - accuracy: 0.9440 - loss: 0.1675 - val_accuracy: 0.9351 - val_loss: 0.1696 Epoch 3/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 57ms/step - accuracy: 0.9478 - loss: 0.1557 Epoch 3: val_accuracy did not improve from 0.93515 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9478 - loss: 0.1556 - val_accuracy: 0.7689 - val_loss: 0.6428 Epoch 4/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 56ms/step - accuracy: 0.9488 - loss: 0.1539 Epoch 4: val_accuracy improved from 0.93515 to 0.94694, saving model to /kaggle/working/model/Model.keras 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9488 - loss: 0.1538 - val_accuracy: 0.9469 - val_loss: 0.1696 Epoch 5/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 56ms/step - accuracy: 0.9492 - loss: 0.1503 Epoch 5: val_accuracy did not improve from 0.94694 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9492 - loss: 0.1503 - val_accuracy: 0.8807 - val_loss: 0.2883 Epoch 6/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 57ms/step - accuracy: 0.9512 - loss: 0.1452 Epoch 6: val_accuracy improved from 0.94694 to 0.95034, saving model to /kaggle/working/model/Model.keras 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 61ms/step - accuracy: 0.9512 - loss: 0.1452 - val_accuracy: 0.9503 - val_loss: 0.1575 Epoch 7/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 56ms/step - accuracy: 0.9517 - loss: 0.1410 Epoch 7: val_accuracy improved from 0.95034 to 0.95374, saving model to /kaggle/working/model/Model.keras 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9517 - loss: 0.1410 - val_accuracy: 0.9537 - val_loss: 0.1498 Epoch 8/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 56ms/step - accuracy: 0.9548 - loss: 0.1345 Epoch 8: val_accuracy improved from 0.95374 to 0.95578, saving model to /kaggle/working/model/Model.keras 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9548 - loss: 0.1345 - val_accuracy: 0.9558 - val_loss: 0.1303 Epoch 9/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 57ms/step - accuracy: 0.9552 - loss: 0.1346 Epoch 9: val_accuracy did not improve from 0.95578 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9552 - loss: 0.1346 - val_accuracy: 0.9145 - val_loss: 0.2288 Epoch 10/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 57ms/step - accuracy: 0.9564 - loss: 0.1301 Epoch 10: val_accuracy did not improve from 0.95578 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9564 - loss: 0.1301 - val_accuracy: 0.9322 - val_loss: 0.1780 Epoch 11/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 56ms/step - accuracy: 0.9549 - loss: 0.1310 Epoch 11: val_accuracy did not improve from 0.95578 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9549 - loss: 0.1310 - val_accuracy: 0.9488 - val_loss: 0.1554 Epoch 12/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 56ms/step - accuracy: 0.9561 - loss: 0.1293 Epoch 12: val_accuracy did not improve from 0.95578 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9561 - loss: 0.1293 - val_accuracy: 0.9070 - val_loss: 0.2489 Epoch 13/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 56ms/step - accuracy: 0.9583 - loss: 0.1271 Epoch 13: val_accuracy did not improve from 0.95578 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9583 - loss: 0.1270 - val_accuracy: 0.9431 - val_loss: 0.1567 Epoch 14/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 57ms/step - accuracy: 0.9585 - loss: 0.1254 Epoch 14: val_accuracy did not improve from 0.95578 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9585 - loss: 0.1254 - val_accuracy: 0.8485 - val_loss: 0.3775 Epoch 15/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 56ms/step - accuracy: 0.9570 - loss: 0.1299 Epoch 15: val_accuracy did not improve from 0.95578 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9570 - loss: 0.1298 - val_accuracy: 0.9215 - val_loss: 0.2048 Epoch 16/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 56ms/step - accuracy: 0.9624 - loss: 0.1178 Epoch 16: val_accuracy did not improve from 0.95578 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9624 - loss: 0.1179 - val_accuracy: 0.9320 - val_loss: 0.1764 Epoch 17/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 56ms/step - accuracy: 0.9582 - loss: 0.1219 Epoch 17: val_accuracy did not improve from 0.95578 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9582 - loss: 0.1218 - val_accuracy: 0.7669 - val_loss: 0.6052 Epoch 18/20 275/276 ━━━━━━━━━━━━━━━━━━━━ 0s 57ms/step - accuracy: 0.9609 - loss: 0.1200 Epoch 18: val_accuracy did not improve from 0.95578 276/276 ━━━━━━━━━━━━━━━━━━━━ 17s 60ms/step - accuracy: 0.9609 - loss: 0.1200 - val_accuracy: 0.9005 - val_loss: 0.2384
Pro trénování jsem použil celou trénovací sadu s tím, že 20% dat jsem vyčlenil pro validaci po každé dávce.
Průběh trénování je možné vidět v následujících grafech:
fig, ax = plt.subplots(1, 2, figsize=(16, 4)) sns.lineplot(data={k: history.history[k] for k in ('loss', 'val_loss')}, ax=ax[0]) sns.lineplot(data={k: history.history[k] for k in history.history.keys() if k not in ('loss', 'val_loss')}, ax=ax[1]) plt.show()
A na závěr si můžu vyzkoušet kvalitu modelu na testovací sadě dat.
Následuje tedy predikce pro testovací sadu. Výsledek je zobrazen jako „Confusion Matrix“.
y_pred = model.predict(x_test, verbose=0) plt.figure(figsize=(6, 3)) heatmap = sns.heatmap(confusion_matrix(np.argmax(y_test, axis=-1), np.argmax(y_pred, axis=-1)), annot=True, fmt="d", cbar=True) heatmap.yaxis.set_ticklabels(LABELS, rotation=90, ha='right') heatmap.xaxis.set_ticklabels(LABELS, rotation=0, ha='right') heatmap.axes.set_ylabel('True label') heatmap.axes.set_xlabel('Predicted label') plt.show()
Doposud tedy stále nic objevného. Výsledek modelu je docela ucházející.
Heatmaps a lokalizace
A nyní se dostávám k podstatě tohoto článku. Pokud udělám predikci na obrázku buňky s infekcí malárie, zajímalo by mne, na základě jaké části obrázku model došel k tomuto závěru. Chtěl bych vidět, která část buňky je považována za infikovanou (jen dovětek, když se na obrázek infikované buňky podíváte, je to hned vidět).
V následujících krocích si tedy vyberu pouze ty vzorky z testovací sady, které jsou infikovány a byly správně klasifikovány (v tomto okamžiku již neřeším kvalitu modelu, pouze interpretují výsledky). Budou mne tedy zajímat pouze vzorky ve třídě 1.
Pro další vyhodnocení budu potřebovat výstup z poslední konvoluční vrstvy modelu. To se jednoduše udělá tak, že si vytvořím nový model pred_model, jehož vstupem bude vstup původního modelu. Výstupem pak bude výstup vrstvy block3_conv1. Tím si zajistím, že budu mít model s trénovanými vrstvami původního modelu. Výstupem jsou pak vlastnosti 16×16×256 (doporučuji podívat se do výpisu modelu výše).
Dále budu potřebovat váhy poslední klasifikační vrstvy pro třídu 1. V mém případě se váhy jmenují last_layer_weights.
PRED_CLASS = 1 pred_model = Model(model.input, model.get_layer('block_3_conv_1').output) last_layer_weights = model.layers[-1].get_weights()[0][:, PRED_CLASS]
Vyberu si tedy náhodně několik vzorků (v tomto případě 10), které byly správně klasifikované jako infikované, a pro každý takovýto vzorek pak postupně tyto kroky:
-
Zobrazím si původní podobu vzorku v prvním sloupci následujícího zobrazení.
-
Pro tento vzorek udělám predikci pomocí modelu pred_model. Následuje dot produkt (omlouvám se, ale naznám český ekvivalent) mezi výsledkem predikce a váhami klasifikační vrstvy. Vznikne mně tak heatmap významnosti jednotlivých částí obrázku. Ještě jsem zde zařadil zvětšení obrázku na původní velikost. Výsledek je zobrazen ve druhém sloupci.
-
V posledním kroku jsem ještě hledal maxima ve dříve spočtené heatmap (maximálně tři na jeden obrázek). Ty mně pak poslouží k namalování čtverečku pro lepší zdůraznění místa infekce v původním obrázku.
Výsledek můžete vidět v následujícím přehledu:
samples = x_test[np.logical_and(np.argmax(y_test, axis=-1) == PRED_CLASS, np.argmax(y_pred, axis=-1) == PRED_CLASS)] rows, cols = 10, 3 fig=plt.figure(figsize=(12, 32)) for i in range(1, rows*cols+1, 3): fig.add_subplot(rows, cols, i) image = samples[random.randrange(samples.shape[0])] plt.imshow(image) fig.add_subplot(rows, cols, i+1) pred_conv = pred_model.predict(np.expand_dims(image, axis=0), verbose=0) heat_map = np.dot(np.squeeze(pred_conv), np.expand_dims(last_layer_weights, axis=-1)) heat_map = cv2.resize(heat_map, IMAGE_SIZE) plt.imshow(cv2.resize(heat_map, IMAGE_SIZE)) fig.add_subplot(rows, cols, i+2) peak_coord = peak_local_max(heat_map, num_peaks=3, min_distance=5, threshold_rel=0.5) for h, w in peak_coord: plt.gca().add_patch(Rectangle((max(w-6, 0), max(h-6, 0)), 12, 12, edgecolor='r', fill=False)) plt.imshow(image) plt.show()
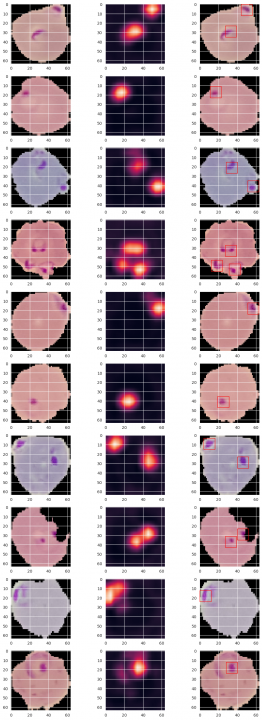
Z příkladů je vidět, že se trefuji do infikovaných míst docela dobře.
Žádné názory

Jiří Raška
pracuje na pozici IT architekta. Poslední roky se zaměřuje na integrační a komunikační projekty ve zdravotnictví. Mezi jeho koníčky patří také paragliding a jízda na horském kole.
Nejčtenější články autora
-
Mravenčí kolonií na Nurikabe
Přečteno 33 870×
-
AutoEncoder na ořechy
Přečteno 30 288×
-
Klasifikace EKG křivek - výlet do světa neuronových sítí
Přečteno 27 558×
-
Detekce anomálií v auditních záznamech - časové řady
Přečteno 25 477×
-
Detekce anomálií v auditních záznamech
Přečteno 20 968×
Poslední názory
-
\
Re: Detectron2 – hra na špióny
ke článku Detectron2 – hra na špióny -
\
Re: Detectron2 – najdi medvěda
ke článku Detectron2 – najdi medvěda -
\
Re: Mravenčí kolonií na Nurikabe
ke článku Mravenčí kolonií na Nurikabe -
\
Re: AutoEncoder na ořechy
ke článku AutoEncoder na ořechy -
\
Re: Rozpoznání zápalu plic z RTG snímků - ViT model
ke článku Rozpoznání zápalu plic z RTG snímků - ViT model