Klasifikace Elektrokardiogramu (EKG) - díl první
Po delší době jsem se vrátil k problému klasifikace EKG křivek s využitím konvoluční neuronové sítě. Tentokrát ovšem na jiné datové sadě, a budu se pokoušet o poněkud odlišný pohled než v předchozím příspěvku Klasifikace EKG křivek – výlet do světa neuronových sítí .
Jedním z prvních problémů, na které člověk narazí při vytváření modelu je, kde vzít nějaká rozumná data. Modely budu vytvářet v prostředí Kaggle, proto jsem nejdříve zamířil do dat poskytovaných na tomto serveru.
Pro své pokusy jsem si vybral datovou sadu PTB-XL ECG dataset.
In [1]:
import sys import os import ast import wfdb import warnings import numpy as np import pandas as pd import matplotlib.pyplot as plt import seaborn as sns import tensorflow as tf import tensorflow.keras as keras import sklearn.metrics from sklearn.preprocessing import StandardScaler sns.set_style('darkgrid')
Jaká data mám k dispozici
Datová sada PTB-XL ECG dataset zahrnuje celkem 21837 klinických vyšetření na 12-ti svodovém EKG přístroji. Celkem bylo vyšetřováno 18885 pacientů. Délka každého vyšetření byla omezena na 10 sekund.
Součástí datové sady jsou nejen vlastní EKG křivky, ale také metadata vztahující se ke každému klinickému vyšetření.
Pokud se podíváte na obsah datové sady, pak metadata o každém vyšetření naleznete v souboru ptbxl_database.csv .
Načtení metadat vyšetření
In [2]:
PATH_TO_DATA = '/kaggle/input/ptb-xl-dataset/ptb-xl-a-large-publicly-available-electrocardiography-dataset-1.0.1/' ECG_df = pd.read_csv(os.path.join(PATH_TO_DATA, 'ptbxl_database.csv'), index_col='ecg_id') ECG_df.scp_codes = ECG_df.scp_codes.apply(lambda x: ast.literal_eval(x)) ECG_df.patient_id = ECG_df.patient_id.astype(int) ECG_df.nurse = ECG_df.nurse.astype('Int64') ECG_df.site = ECG_df.site.astype('Int64') ECG_df.validated_by = ECG_df.validated_by.astype('Int64') ECG_df
Out[2]:
|
patient_id |
age |
sex |
height |
weight |
nurse |
site |
device |
recording_date |
report |
… |
validated_by_human |
baseline_drift |
static_noise |
burst_noise |
electrodes_problems |
extra_beats |
pacemaker |
strat_fold |
filename_lr |
filename_hr |
|
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
ecg_id |
|||||||||||||||||||||
|
1 |
15709 |
56.0 |
1 |
NaN |
63.0 |
2 |
0 |
CS-12 E |
1984–11–09 09:17:34 |
sinusrhythmus periphere niederspannung |
… |
True |
NaN |
, I-V1, |
NaN |
NaN |
NaN |
NaN |
3 |
records100/00000/00001_lr |
records500/00000/00001_hr |
|
2 |
13243 |
19.0 |
0 |
NaN |
70.0 |
2 |
0 |
CS-12 E |
1984–11–14 12:55:37 |
sinusbradykardie sonst normales ekg |
… |
True |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
2 |
records100/00000/00002_lr |
records500/00000/00002_hr |
|
3 |
20372 |
37.0 |
1 |
NaN |
69.0 |
2 |
0 |
CS-12 E |
1984–11–15 12:49:10 |
sinusrhythmus normales ekg |
… |
True |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
5 |
records100/00000/00003_lr |
records500/00000/00003_hr |
|
4 |
17014 |
24.0 |
0 |
NaN |
82.0 |
2 |
0 |
CS-12 E |
1984–11–15 13:44:57 |
sinusrhythmus normales ekg |
… |
True |
, II,III,AVF |
NaN |
NaN |
NaN |
NaN |
NaN |
3 |
records100/00000/00004_lr |
records500/00000/00004_hr |
|
5 |
17448 |
19.0 |
1 |
NaN |
70.0 |
2 |
0 |
CS-12 E |
1984–11–17 10:43:15 |
sinusrhythmus normales ekg |
… |
True |
, III,AVR,AVF |
NaN |
NaN |
NaN |
NaN |
NaN |
4 |
records100/00000/00005_lr |
records500/00000/00005_hr |
|
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
… |
|
21833 |
17180 |
67.0 |
1 |
NaN |
NaN |
1 |
2 |
AT-60 3 |
2001–05–31 09:14:35 |
ventrikulÄre extrasystole(n) sinustachykardie … |
… |
True |
NaN |
, alles, |
NaN |
NaN |
1ES |
NaN |
7 |
records100/21000/21833_lr |
records500/21000/21833_hr |
|
21834 |
20703 |
93.0 |
0 |
NaN |
NaN |
1 |
2 |
AT-60 3 |
2001–06–05 11:33:39 |
sinusrhythmus lagetyp normal qrs(t) abnorm … |
… |
True |
NaN |
NaN |
NaN |
NaN |
NaN |
NaN |
4 |
records100/21000/21834_lr |
records500/21000/21834_hr |
|
21835 |
19311 |
59.0 |
1 |
NaN |
NaN |
1 |
2 |
AT-60 3 |
2001–06–08 10:30:27 |
sinusrhythmus lagetyp normal t abnorm in anter… |
… |
True |
NaN |
, I-AVR, |
NaN |
NaN |
NaN |
NaN |
2 |
records100/21000/21835_lr |
records500/21000/21835_hr |
|
21836 |
8873 |
64.0 |
1 |
NaN |
NaN |
1 |
2 |
AT-60 3 |
2001–06–09 18:21:49 |
supraventrikulÄre extrasystole(n) sinusrhythmu… |
… |
True |
NaN |
NaN |
NaN |
NaN |
SVES |
NaN |
8 |
records100/21000/21836_lr |
records500/21000/21836_hr |
|
21837 |
11744 |
68.0 |
0 |
NaN |
NaN |
1 |
2 |
AT-60 3 |
2001–06–11 16:43:01 |
sinusrhythmus p-sinistrocardiale lagetyp norma… |
… |
True |
NaN |
, I-AVL, |
NaN |
NaN |
NaN |
NaN |
9 |
records100/21000/21837_lr |
records500/21000/21837_hr |
21837 rows × 27 columns
Když se podíváte detailněji na záznamy o vyšetření, pak tam najdete informace týkající se samotného pacienta. Dále pak podmínky, za kterých bylo vyšetření provedeno a popsáno. No a nakonec také odkaz na adresáře, ve kterých jsou uložena data vlastních křivek.
Navíc autoři datové sady navrhli rozdělení vyšetření do skupin. Jedná se celkem o 10 skupin. Autoři předpokládají, že prvních osm skupin bude použito pro trénování, devátá skupina bude použita jako validační, a poslední skupina jako testovací. Budu se tohoto doporučení držet.
In [3]:
ECG_df.strat_fold.value_counts()
Out[3]:
strat_fold 10 2203 3 2194 9 2193 2 2184 8 2179 7 2178 6 2178 1 2177 5 2176 4 2175 Name: count, dtype: int64
Ještě mně chybí přiřazení diagnóz k jednotlivým EKG vyšetřením. S nimi je to poněkud komplikovanější. V datové saděECG_df mám sloupec scp_codes , který obsahuje slovník kódů a k nim přiřazených pravděpodobností výskytu. Tyto kódy pak odkazují do druhé tabulky, která je uložena v souboru stc_statements.csv .
Pro další práci budu tedy potřebovat i tuto tabulku:
In [4]:
SCP_df = pd.read_csv(os.path.join(PATH_TO_DATA, 'scp_statements.csv'), index_col=0)
SCP_df = SCP_df[SCP_df.diagnostic == 1]
Ze všech definovaných kódů jsem vybral pouze ty spadající do kategorie diagnostických.
Dále potřebuji doplnit kódy diagnostických tříd do datové sady ECG_df , a to propojením na datovou saduSCP_df přes sloupec scp_codes .
In [5]:
def diagnostic_class(scp):
res = set()
for k in scp.keys():
if k in SCP_df.index:
res.add(SCP_df.loc[k].diagnostic_class)
return list(res)
ECG_df['scp_classes'] = ECG_df.scp_codes.apply(diagnostic_class)
V datové sadě mně přibyl sloupec scp_classes , který obsahuje seznam diagnostických tříd přiřazených k danému vyšetření. Jedná se skutečně o seznam, což tedy znamená, že jedno vyšetřením může mít přiřazeno více diagnostických tříd. Toto je pro další část textu velice podstatná informace.
Takto vypadá přehled definovaných diagnostických tříd:
Records | Superclass | Description9528 | NORM | Normal ECG5486 | MI | Myocardial Infarction5250 | STTC | ST/T Change4907 | CD | Conduction Disturbance2655 | HYP | Hypertrophy
A dále se můžeme podívat na zastoupení diagnostických tříd v celé datové sadě:
In [6]:
ECG_df.scp_classes.value_counts()
Out[6]:
scp_classes [NORM] 9083 [MI] 2538 [STTC] 2406 [CD] 1709 [MI, CD] 1302 [STTC, HYP] 783 [MI, STTC] 602 [HYP] 536 [STTC, CD] 472 [] 407 [NORM, CD] 407 [MI, STTC, HYP] 362 [CD, HYP] 300 [MI, STTC, CD] 223 [STTC, CD, HYP] 211 [MI, HYP] 183 [MI, STTC, CD, HYP] 158 [MI, CD, HYP] 117 [NORM, STTC] 28 [NORM, STTC, CD] 5 [NORM, CD, HYP] 2 [NORM, HYP] 2 [MI, NORM, CD, HYP] 1 Name: count, dtype: int64
Načtení EKG křivek
Jednotlivé křivky jsou uloženy v souborech ve formátu WaveForm DataBase (WFDB), a to ve dvou variantách. Vždy se jedná o 10-ti sekundové záznamy s vzorkovací frekvencí 100Hz nebo 500Hz. Vzhledem k potřebě redukovat objem dat, se kterými budu dále pracovat, jsem si vybral tu menší frekvenci.
Takto si tedy načtu data křivek:
In [7]:
def load_raw_data(df, sampling_rate, path):
if sampling_rate == 100:
data = [wfdb.rdsamp(os.path.join(path, f)) for f in df.filename_lr]
else:
data = [wfdb.rdsamp(os.path.join(path, f)) for f in df.filename_hr]
data = np.array([signal for signal, meta in data])
return data
sampling_rate = 100
ECG_data = load_raw_data(ECG_df, sampling_rate, PATH_TO_DATA)
ECG_data.shape
Out[7]:
(21837, 1000, 12)
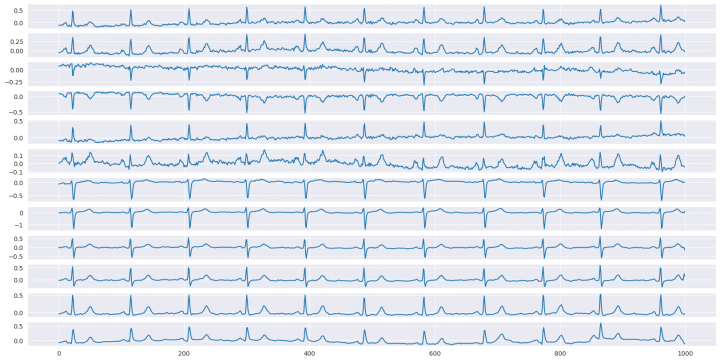
Z dimenzí datové sady ECG_data je zřejmé, že se mně podařilo načíst 21837 vzorků. Každý vzorek obsahuje 1000 hodnot křivky (10 sekund * 100Hz vzorkování) pro 12 EKG svodů.
Jen pro představu, takto vypadá jeden vzorek dat:
In [8]:
sample = ECG_data[0] bar, axes = plt.subplots(sample.shape[1], 1, figsize=(20,10)) for i in range(sample.shape[1]): sns.lineplot(x=np.arange(sample.shape[0]), y=sample[:, i], ax=axes[i]) plt.show()
A ještě jeden pohled na metadata vyšetření
Zcela jistě stojí za úvahu pohled na kvalitu dat (v tomto případě mám na mysli především metadata), a to s ohledem jejich zařazení do dalšího procesu modelování.
Takto vypadá zevrubný pohled na datovou saduECG_df z hlediska nedefinovaných hodnot:
In [9]:
import missingno as msno msno.matrix(ECG_df) plt.show()

A takto vypadá pohled na jednotlivé sloupce z hlediska počtu unikátních hodnot v nich:
In [10]:
ECG_df[[col for col in ECG_df.columns if col not in ('scp_codes', 'scp_classes')]].nunique(dropna=True)
Out[10]:
patient_id 18885 age 94 sex 2 height 77 weight 127 nurse 12 site 51 device 11 recording_date 21813 report 9883 heart_axis 8 infarction_stadium1 6 infarction_stadium2 3 validated_by 12 second_opinion 2 initial_autogenerated_report 2 validated_by_human 2 baseline_drift 321 static_noise 124 burst_noise 103 electrodes_problems 14 extra_beats 128 pacemaker 4 strat_fold 10 filename_lr 21837 filename_hr 21837 dtype: int64
Z výše uvedeného je zřejmé, že nemá velký význam zahrnovat některé sloupce do dalšího modelování, neboť jejich vliv na výsledek by byl malý.
Příprava dat pro modelování
Datová sada X – metadata
Prvním zdrojem informací, které mohu zahrnout to modelování, jsou data o pacientech a podmínkách provedeného EKG vyšetření. Jedná se o data v tabulce ECG_df , a již dříve jsem je označoval jako metadata.
Z výše naznačeného pohledu na metadata je zřejmé, že do modelování nebudu zahrnovat všechny údaje uvedené ve zdrojové tabulce. Vyberu si pouze ty údaje, které se nějakým způsobem vztahují k vyšetřovanému pacientovi a jeho zdravotním stavu.
Výsledkem pak bude datová sada X , která mně bude sloužit jako jeden z možných vstupů do modelování.
In [11]:
X = pd.DataFrame(index=ECG_df.index) X['age'] = ECG_df.age X.age.fillna(0, inplace=True) X['sex'] = ECG_df.sex.astype(float) X.sex.fillna(0, inplace=True) X['height'] = ECG_df.height X.loc[X.height < 50, 'height'] = np.nan X.height.fillna(0, inplace=True) X['weight'] = ECG_df.weight X.weight.fillna(0, inplace=True) X['infarction_stadium1'] = ECG_df.infarction_stadium1.replace({ 'unknown': 0, 'Stadium I': 1, 'Stadium I-II': 2, 'Stadium II': 3, 'Stadium II-III': 4, 'Stadium III': 5 }).fillna(0) X['infarction_stadium2'] = ECG_df.infarction_stadium2.replace({ 'unknown': 0, 'Stadium I': 1, 'Stadium II': 2, 'Stadium III': 3 }).fillna(0) X['pacemaker'] = (ECG_df.pacemaker == 'ja, pacemaker').astype(float) X
Out[11]:
|
age |
sex |
height |
weight |
infarction_stadium1 |
infarction_stadium2 |
pacemaker |
|
|---|---|---|---|---|---|---|---|
|
ecg_id |
|||||||
|
1 |
56.0 |
1.0 |
0.0 |
63.0 |
0.0 |
0.0 |
0.0 |
|
2 |
19.0 |
0.0 |
0.0 |
70.0 |
0.0 |
0.0 |
0.0 |
|
3 |
37.0 |
1.0 |
0.0 |
69.0 |
0.0 |
0.0 |
0.0 |
|
4 |
24.0 |
0.0 |
0.0 |
82.0 |
0.0 |
0.0 |
0.0 |
|
5 |
19.0 |
1.0 |
0.0 |
70.0 |
0.0 |
0.0 |
0.0 |
|
… |
… |
… |
… |
… |
… |
… |
… |
|
21833 |
67.0 |
1.0 |
0.0 |
0.0 |
0.0 |
0.0 |
0.0 |
|
21834 |
93.0 |
0.0 |
0.0 |
0.0 |
4.0 |
0.0 |
0.0 |
|
21835 |
59.0 |
1.0 |
0.0 |
0.0 |
0.0 |
0.0 |
0.0 |
|
21836 |
64.0 |
1.0 |
0.0 |
0.0 |
0.0 |
0.0 |
0.0 |
|
21837 |
68.0 |
0.0 |
0.0 |
0.0 |
0.0 |
0.0 |
0.0 |
21837 rows × 7 columns
Datová sada Y – EKG křivky
Datová sada Y mně bude představovat hlavním zdroj vstupních informací do modelování, tedy naměřené EKG křivky.
TabulkaECG_data plně postačuje pro tento účel, takže v tomto okamžiku mám již hotovo.
In [12]:
Y = ECG_data
Y.shape
Out[12]:
(21837, 1000, 12)
Datová sada Z – klasifikační třídy
V tomto případě se jedná o cílové hodnoty, které chceme zjišťovat. Jinak řečeno, v datové saděZ jsou informace o zařazení každého vyšetření do diagnostických tříd.
Sloupce v datové saděZ odpovídají názvům klasifikačních tříd. Zařazení/nezařazení vyšetření do třídy se pak určuje hodnotou 1/0.
In [13]:
Z = pd.DataFrame(0, index=ECG_df.index, columns=['NORM', 'MI', 'STTC', 'CD', 'HYP'], dtype='int') for i in Z.index: for k in ECG_df.loc[i].scp_classes: Z.loc[i, k] = 1 Z
Out[13]:
|
NORM |
MI |
STTC |
CD |
HYP |
|
|---|---|---|---|---|---|
|
ecg_id |
|||||
|
1 |
1 |
0 |
0 |
0 |
0 |
|
2 |
1 |
0 |
0 |
0 |
0 |
|
3 |
1 |
0 |
0 |
0 |
0 |
|
4 |
1 |
0 |
0 |
0 |
0 |
|
5 |
1 |
0 |
0 |
0 |
0 |
|
… |
… |
… |
… |
… |
… |
|
21833 |
0 |
0 |
1 |
0 |
0 |
|
21834 |
1 |
0 |
0 |
0 |
0 |
|
21835 |
0 |
0 |
1 |
0 |
0 |
|
21836 |
1 |
0 |
0 |
0 |
0 |
|
21837 |
1 |
0 |
0 |
0 |
0 |
21837 rows × 5 columns
Jen pro představu o zastoupení jednotlivých tříd v celé datové sadě si můžeme ukázat následující:
In [14]:
Z.sum()
Out[14]:
NORM 9528 MI 5486 STTC 5250 CD 4907 HYP 2655 dtype: int64
A tady můžete vidět jeden z problémů, který sebou tato datová sada nese, a sice nerovnoměrné zastoupení vzorků s různými třídami. Jak tomuto problému čelit bude možná někdy v budoucnu námětem pro další bádání, ale v tomto příspěvku to nebude.
Rozdělení datových sad na trénovací, validační a testovací
Posledním krokem v přípravě dat bude jejich rozdělení na podmnožiny určené pro trénování sítě, validaci a ověření výsledku. Budu respektovat doporučení autorů datové sady, a pro rozdělení použiji atribut ECG_df.strat_fold .
In [15]:
X_train, Y_train, Z_train = X[ECG_df.strat_fold <= 8], ECG_data[X[ECG_df.strat_fold <= 8].index - 1], Z[ECG_df.strat_fold <= 8] X_valid, Y_valid, Z_valid = X[ECG_df.strat_fold == 9], ECG_data[X[ECG_df.strat_fold == 9].index - 1], Z[ECG_df.strat_fold == 9] X_test, Y_test, Z_test = X[ECG_df.strat_fold == 10], ECG_data[X[ECG_df.strat_fold == 10].index - 1], Z[ECG_df.strat_fold == 10] print(f"Trénovací sady: X_train={X_train.shape} Y_train={Y_train.shape} Z_train={Z_train.shape}") print(f"Validační sady: X_valid={X_valid.shape} Y_valid={Y_valid.shape} Z_valid={Z_valid.shape}") print(f"Testovací sady: X_test ={X_test.shape} Y_test={Y_test.shape} Z_test={Z_test.shape}") Trénovací sady: X_train=(17441, 7) Y_train=(17441, 1000, 12) Z_train=(17441, 5) Validační sady: X_valid=(2193, 7) Y_valid=(2193, 1000, 12) Z_valid=(2193, 5) Testovací sady: X_test =(2203, 7) Y_test=(2203, 1000, 12) Z_test=(2203, 5)
Datová sada S – Spektrogramy EKG křivek
Doposud jsem se zabýval výstupy EKG křivek z jednotlivých elektrod tak, jak je poskytují tvůrci datové sady PTB-XL ECG dataset. Jedná se tedy o pohled v časové rovině (připomínám že signály mají délku 10 sekund).
Při analýze signálů se velmi často využívá ještě jiný pohled, a sice jeho spektrum. V případě EKG křivek obvykle budu hledat nějaké anomálie v jejich průběhu, což by se mělo projevit ve změně spektra v inkriminované oblasti křivky. Nevystačím si tedy s pouhým převedením signálu na jeho spektrum. Potřebuji propojit oba pohledy, a to jak ve spektrální rovině, tak také v časové. Jinými slovy potřebuji vytvořit spektrogram (short-time Fourier transform, STFT).
Pro výpočet spektrogramu jsem použil knihovnu librosa.
Nejdříve si potřebuji upravit pořadí dimenzí v datovém setuEVG_data tak, aby svody byly ve druhém rozměru, a křivky v tom posledním.
In [16]:
ECG_swapped = np.swapaxes(ECG_data, 1, 2)
ECG_swapped.shape
Out[16]:
(21837, 12, 1000)
A nyní si vytvořím spektrum pomocí funkce pro STFT. Parametry funkce jsem zvolil tak, abych měl přibližně stejný počet rámců jak v časové, tak se spektrální rovině.
Výsledek je ještě upraven přepočtem amplitudy na výkon a převeden na decibely(dB).
import librosaframe_size = 60hop_size = 30S = librosa.stft(ECG_swapped, n_fft=frame_size, hop_length=hop_size)S = np.abs(S) ** 2S = librosa.power_to_db(S)S.shape
Tato část kódu je bohužel hodně paměťově náročná, nebylo možné ji v prostředí Kaggle spustit. Proto jsem tento výpočet udělal na jiném stroji a zde si jen načtu výsledek …
In [17]:
PREPROCESSED_DATA_FILE = '/kaggle/input/ptb-xl-preprocessed-dataset/data.npz' thismodule = sys.modules[__name__] with np.load(PREPROCESSED_DATA_FILE) as data: for k in ('S_train', 'S_valid', 'S_test'): setattr(thismodule, k, data[k].astype(float)) print(f"S_train={S_train.shape} S_valid={S_valid.shape} S_test={S_test.shape}") S_train=(17441, 12, 31, 34) S_valid=(2193, 12, 31, 34) S_test=(2203, 12, 31, 34)
Spektrogramy mám již rozdělené do tří sad pro trénování, validaci a testování.
Z výše vypsaných dimenzí je zřejmé, že u každého vzorku mám pro každý svod spektrogram s rozlišením 31 hodnot ve spektrální rovině a 34 hodnot v rovině časové.
Pro představu si zkusím jeden takový spektrogram zobrazit:
In [18]:
import librosa frame_size = 60 hop_size = 30 def plot_spectrogram(y, sr, hop_length, y_axis = "linear"): plt.figure(figsize = (8, 4)) librosa.display.specshow(y, sr = sr, hop_length = hop_length, x_axis = "time", y_axis = y_axis) plt.colorbar(format="%+2.f") plot_spectrogram(S_train[0, 0], sr=frame_size, hop_length=hop_size)

A to je pro dnešek vše. Ve druhém dílu budu pokračovat vlastními modely zdrojových dat.

Jiří Raška
pracuje na pozici IT architekta. Poslední roky se zaměřuje na integrační a komunikační projekty ve zdravotnictví. Mezi jeho koníčky patří také paragliding a jízda na horském kole.
Nejčtenější články autora
-
Mravenčí kolonií na Nurikabe
Přečteno 33 835×
-
AutoEncoder na ořechy
Přečteno 30 256×
-
Klasifikace EKG křivek - výlet do světa neuronových sítí
Přečteno 27 514×
-
Detekce anomálií v auditních záznamech - časové řady
Přečteno 25 429×
-
Detekce anomálií v auditních záznamech
Přečteno 20 937×
Poslední názory
-
\
Re: Detectron2 – hra na špióny
ke článku Detectron2 – hra na špióny -
\
Re: Detectron2 – najdi medvěda
ke článku Detectron2 – najdi medvěda -
\
Re: Mravenčí kolonií na Nurikabe
ke článku Mravenčí kolonií na Nurikabe -
\
Re: AutoEncoder na ořechy
ke článku AutoEncoder na ořechy -
\
Re: Rozpoznání zápalu plic z RTG snímků - ViT model
ke článku Rozpoznání zápalu plic z RTG snímků - ViT model



















